在2013年底,Nature Method 期刊將單細胞定序選為年度技術。在發育生物學、腫瘤基因體學、生殖醫學等領域,單細胞定序技術已經開始發揮它的威力,為我們帶來許多珍貴的訊息。本篇主要著重於此技術在當前的現況,介紹目前的單細胞分離技術、放大技術、與一些應用的面向。第一部分將先介紹技術的部份,第二部分將整理一些已發表的應用情形。
單細胞定序的流程,大概可分為1. 單細胞分離,2.依照不同的需求放大樣本進行定序。可以參考圖一。
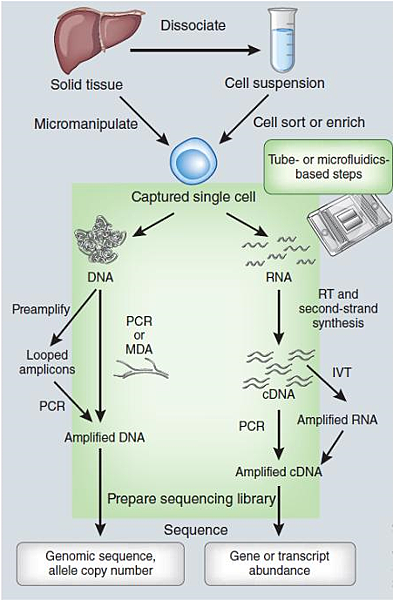
圖一 單細胞定序的流程(圖片來源: Single-cell sequencing Vol.11 No.1 | JAN 2014 |Nature Methods)
單細胞分離技術
一般來說,多細胞物種很少有均質的組織,總是由許多的細胞類型所組成。要從這些細胞中將我們想要的一顆細胞分離出來,並不是件容易的事情。先要將組織從動物或植物中取下,接著將整個組織分離成一顆顆的細胞(通常是用酵素來分開細胞)。然後將單一顆細胞分隔開,個別水解並進行後續實驗。而將單一顆細胞分隔開的方法大概有如表一的種類,各有其優缺點。圖二則為各種方法的示意圖。

(表來源:Shapiro E, Biezuner T, Linnarsson S. 2013. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet 14:618–630.)

圖二 各式單細胞分離方法(圖片來源:Navin, Nicholas, and James Hicks. "Future Medical Applications of Single-cell Sequencing in Cancer." Genome Medicine 3.5 (2011): 31.)
顯微操作法(micromanipulation method)的優點是比較簡單且便宜,缺點在於只能使用在懸浮的細胞。比較低的產量,且容易因為辨識細胞困難等原因出錯。
雷射顯微擷取系統(Laser-capture microdissection)則可應用於固定的組織或是冷凍切片上,而且可以得知細胞在組織之中的位置資訊。缺點主要在於低產量、操作相對困難,且不容易得到完整的核酸或是容易被其他細胞的核酸汙染。
流式細胞儀細胞分選法可以快速且大量的篩選細胞,而且可以藉由免疫方法進行細胞標定,進而篩選出特定細胞。不過由於需要大量起始細胞來進行篩選,且快速的液流系統有可能會傷害到細胞。在讓細胞懸浮的過程之中也有可能會改變細胞的特性,進而讓細胞內部的transcriptome改變。
單細胞基因體放大技術的方法與限制
一個雙套染色體的人類細胞大概有7pg的染色體DNA,需要進一步放大才足夠現有的定序技術進行Library製備。目前現行的WGA(Whole-genome Amplification)放大方法有MDA(multiple displacement amplification)、PCR為基礎的方法、MALBAC(Multiple Annealing and Looping Based Amplification Cycles)。
PCR方法主要有三種,Linker Adapter PCR(LA-PCR),將Adapter接在合適大小的DNA片段上後,進行PCR放大的方法,如圖三所示。此方法的效果取決於片段接合adapter的效果與放大時候偏誤情形的嚴重程度。

圖三 Linker Adapter PCR(LA-PCR) (圖片來源: http://www.sigmaaldrich.com/technical-documents/articles/life-science-innovations/whole-genome-amplification.html#ref)
Degenerative oligonucleotide primer (DOP) PCR,此方法是使用部分一組oligo具有隨機的3’端與部分固定的5’端,放大的過程中一開始先使用較低的annealing溫度,再使用高的annealing溫度進行PCR。此方法需要良好的primer設計,讓其可以很平均的黏合在DNA序列之上。

圖四 Degenerative oligonucleotide primer (DOP) PCR
(圖片來源:http://www.sigmaaldrich.com/technical-documents/articles/life-science-innovations/whole-genome-amplification.html#ref)
Primer extension pre-amplification (PEP) PCR使用random hexamer,先使用非常低的annealing溫度去進行數個cycle,再增加annealing溫度去進行後續放大。
此方法的Uniformity常常不是很好,且其缺點大部分可以被MDA方法解決,目前多使用MDA方法。

圖五 MDA(multiple displacement amplification) (圖片來源: http://en.wikipedia.org/wiki/File:Single_cell_sequencing_%EF%BC%88MDA%EF%BC%89.JPG)
MDA法使用了Random primer 與Phi 29 polymerase,可將一顆細胞的genomic DNA放大到約1~2μg相關流程如圖。由於Phi29具有displacement的能力,MDA會產生一個多分支DNA產物,使放大產物能夠有足夠的覆蓋率。另外因Phi 29是high fidelity的polymerase,由於錯誤的狀況少,可以使用在SNP的偵測之中。
2012年,Zong等人在Science發表了MALBAC法。由於Primer的特殊設計,此法可產生形成環狀的amplicom。一開始先進行數個循環的線性放大,然後再進行PCR放大。藉由此法,Zong等人由單顆細胞可得到93%的覆蓋率的人類基因體,並且偵測到單顆細胞的Copy number variation。此法的優點在於可由pg等級的DNA開始,由單顆細胞來進行放大。較高的覆蓋率,較低的放大偏誤,與MDA比較,MALBAC有較低的Allele drop out(1%)而MDA可高到65%,在SNP的偵測效率上,也較MDA高。但由於使用的是error-prone的polymerase,可能會因此產生較高的錯誤,也因此可能需要定序2~3顆細胞來確認結果。

圖六 MALBAC(Multiple Annealing and Looping Based Amplification Cycles)(圖片來源: http://en.wikipedia.org/wiki/File:Multiple_Annealing_Looped_Base_Amplification_Cycles_(MALBAC)_Workflow.jpg)
Single-Cell Transcriptomics
前面介紹了關於單細胞基因體擴增的方法,接著來介紹單細胞的RNA擴增技術。
人類細胞可能只有少於1 pg的mRNA,而且表現量在不同細胞之中的差異很大,有許多基因(可多到85%的表現基因)在細胞中只表現100 copies以下(如圖七所示)。每個細胞的轉錄體表現也不是固定的,會隨著細胞本身的週期與環境狀態做改變。此外,mRNA的生合成與分解也會有許多變化,造成個別細胞的差異性很大。這些都增加了分析的困難度。

圖七 mRNA transcript表現量示意圖 (圖片來源: Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)
RNA擴增技術
最早的單細胞轉錄體放大技術是in vitro transcription(IVT),此技術與Illumina定序系統結合,就是圖八所示cell expression by linear amplification and sequencing(CEL-Seq)方法。此方法的優點為使用線性放大的方式,不會有指數放大如PCR所造成的偏誤。缺點在於library會多為基因3’端的部分。

圖八Cell expression by linear amplification and sequencing(CEL-Seq) (圖片來源: Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)
Tang等人的方法如圖九是在第一股cDNA上加上polyA tail,並藉由含有Oligo dT的primer進行第二股cDNA的合成,接著進行PCR放大。此方法與CEL-seq法有類似的問題,library會比較偏向在3’端。

圖九 Tang et al. Method (圖片來源:Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)
模板轉換(Template Switch)的方法主要是利用Moloney murine leukaemia virus family的reverse transcriptases多半會在第一段cDNA上加入小片段的cytosine,藉著helper oligonucleotide上帶有GGG motif,接合cytosine,reverse transcriptase會轉換模板,因而將helper oligonucleotide的片段接到template上。STRT法(圖十)與SMARTer法(圖十一)皆是利用模板轉換的原理來進行修改的替代方法。

圖十 STRT法(圖片來源:Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)

圖十一SMARTer 法(圖片來源:Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)
圖十二針對上述介紹的各種各種方法所針對的定序區域做成了示意圖,顯示各種方法所做的library的涵蓋範圍與特色。

圖十二 (圖片來源:Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126.)
Reference:
- 1. Single-cell sequencing Vol.11 No.1 | JAN 2014 |Nature Methods
- 2. Macaulay IC, Voet T: Single cell genomics: advances and future perspectives. PLoS Genet 2014, 10:e1004126
- 3. Shapiro E, Biezuner T, Linnarsson S. 2013. Single-cell sequencing-based technologies will revolutionize whole-organism science. Nat Rev Genet 14:618–630
- 4. Navin, Nicholas, and James Hicks. "Future Medical Applications of Single-cell Sequencing in Cancer." Genome Medicine 3.5 (2011): 31.
- 5. http://www.sigmaaldrich.com/technical-documents/articles/life-science-innovations/whole-genome-amplification.html#ref
- 6. http://en.wikipedia.org/wiki/File:Single_cell_sequencing_%EF%BC%88MDA%EF%BC%89.JPG
- 7. http://en.wikipedia.org/wiki/File:Multiple_Annealing_Looped_Base_Amplification_Cycles_(MALBAC)_Workflow.jpg





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
