作者:洪郁豪/有勁生物科技
NGS次世代定序技術近年以其高正確率、高輸出量的優勢,刺激了總體基因體定序(Metagenomics Sequencing)技術以微生物相為對象的研究發展。國外的人體微生物相研究大型計畫─Human Microbiome Project,便是利用次世代定序技術,挑選16S rRNA中具鑑別度的部分區域進行定序,然後將大量的數據分析結果,與多種人類疾病做關聯,是為這類研究計畫的標竿之一。其他諸如糖尿病、炎症性腸病、自閉症等疾病研究,也都被陸續報導指出與人體微生物相息息相關。
微生物相的總體基因體定序因受限於序列的讀長,主流上仍會挑選16S rRNA中V1到V9中的某段高度變異區域作為研究工具;但單靠這份資訊,能否正確作出物種分類鑑定,並進一步剖析結果至種(species)的層級,仍值得深究。為了改善上述次世代定序技術的短讀長問題,利用第三代定序平台來解序16S rRNA的全長序列是個有潛力的變通方法。三代定序系統平台中的Nanopore (MinION)具有成本較低、方便性高、定序速度快的優勢,縱使結果的平均正確率只有80%左右,但用此平台定序取得的相關基因研究學術文獻卻持續增多。本文要介紹的研究1,便是將三代定序系統的Nanopore與次世代定序系統也常用於16S rRNA研究的Illumina Miseq,針對其序列讀長不同以及資料輸出量高低差異進行比較,並論述個別分析的結果。
這個研究是以老鼠排泄物做為實驗材料,將排泄物菌相的16S rRNA,分別以Miseq和Nanopore去針對V3-V4區域(前者)和全長序列(後者)進行定序分析。觀察下圖一的定序品質分布,可見到Illumina系統資訊以Q20做reads的品質篩選,而Nanopore的結果則大多落在Q9.7上下;至於讀長的部分,Illumina資料經序列整合(merge)後,定序長度平均為447 bp,而Nanopore的結果則達1393 bp上下,與16S rRNA全長相當接近。
圖一、Illumina與Nanopore定序資料的品質總分(quality score)以及定序長度分布圖

圖片來源:Shin J, Lee S, Go MJ, et al. Analysis of the mouse gut micro-biome using full-length 16S rRNA amplicon sequencing. Sci Rep. 2016 Jul 14; 6:29681-29691.
正確率方面,由Nanopore的內部對照組(internal control)結果可得知本次結果正確率為79.6%,與一般認知其擁有的80%平均正確率近似。物種分類鑑定可達層級方面,該研究將Miseq取得的V3-V4短reads,經Quality trim後再用QIIME pipeline做OTU分析,隨後並經RDP classifier比對Greengenes ( http://greengenes.secondgenome.com/)資料庫進行分群;而Nanopore部分,則將取得的全長序列長reads用LAST aligner v.658比對Greengenes資料庫進行分群處理。從分析結果來看,兩種定序平台均可鑑定出同樣的8門13綱細菌 (見下圖二);倘進一步以heat map方式來呈現與比較兩種平台在鑑別目、科、屬、種等層級菌種的表現差異(下圖三),巨觀來看,兩平台鑑定優勢物種的結果在此處有九分相似(89%)。利用Nanopore定序能鑑別出的小鼠腸內道微生物種類,都與過去各文獻中曾被報導過的鑑定結果相似,顯示Nanopore在16S rRNA定序鑑定菌種方面,也具有很不錯的可信賴度。
圖二、Illumina與Nanopore分析資料在門與綱層級的交叉比對結果
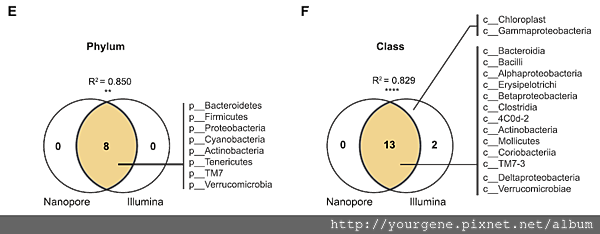
圖片來源:Shin J, Lee S, Go MJ, et al. Analysis of the mouse gut micro-biome using full-length 16S rRNA amplicon sequencing. Sci Rep. 2016 Jul 14; 6:29681-29691.
那麼,從Nanopore的定序結果能否得知額外的訊息呢?從圖三的heatmap結果可見,B圖中最右邊種(Species)的兩平台分析結果與前面目科屬明顯不同。在種這個層級被鑑定出的菌種,Nanopore和Illumina Miseq兩平台都有鑑定到的第I類和只被Illumina Miseq找出來的第III類,都比其在目、科、屬層級所鑑定出的菌種來得少;唯有Nanopore平台在種這個層級有辦法取得比在目科屬更亮眼的鑑定結果。這個在「種」階層出現的分類差異或可歸究平台間讀長及定序深度不同所造成。若進一步在Nanopore定序資料中仔細觀察,我們還可以找到Illumina平台定序結果中沒出現的Bifidobacterium animalis 與Bifidobacterium pseudolongum 兩屬細菌,然而這兩屬菌在過去文獻報導裡,其實都可說是小鼠腸內道的指標菌株。由此研究推論,假如能夠取得近乎全長的rRNA序列,就有機會將菌相分類進一步細分至種的層級。
根據以上研究論述,加上Nanopore定序平台快速、價低、方便的優點,以及一直以來在分子診斷醫學上被認可的潛力,即使尚無法撼動目前NGS在這領域的優勢,仍能為將來開展出許多新的可能性。不過Nanopore系統仍受限於其正確率與定序品質未能提高,或與二代機種相較起來仍顯昂貴的現況,而未能普遍;未來隨著新一代試劑版本陸續問世,或許就有機會克服這些問題。
圖三、Heatmap圖呈現從目到種四個分類層級的Illumina與Nanopore分析資料交叉比對結果

B圖依據兩種平台的鑑定結果,將取得的菌種分成三類;第I類是兩平台都有鑑定到的菌種,第II類是只被Nanopore平台鑑定出的菌種,第III類是只被Illumina Miseq平台鑑定出的菌種。圖片來源:Shin J, Lee S, Go MJ, et al. Analysis of the mouse gut micro-biome using full-length 16S rRNA amplicon sequencing. Sci Rep. 2016 Jul 14; 6:29681-29691.
參考文獻
1. Shin J, Lee S, Go MJ, et al. Analysis of the mouse gut micro-biome using full-length 16S rRNA amplicon sequencing. Sci Rep. 2016 Jul 14; 6:29681-29691. DOI:10.1038/srep29681





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
