Metagenomics這十年來才發展出來的新學門,主要是透過研究分析環境基因體,進而來描繪環境的代謝特性。
一個完整的環境系統,是由許多種生物組成,這些生物種類包羅萬象,除了肉眼可見的動植物外,還有肉眼看不見的微生物族群,這個族群數量遠比動植物多,包含真核微生物(Eukaryotic microbes)、細菌(Bacteria)、古生菌(Archaea)以及病毒(Virus),然而,百分之99以上的微生物是無法培養的,欲描繪一個真實環境的生化代謝圖譜,最有效的方式就是直接萃取環境中的DNA或RNA加以分析,藉由這些基因體資訊,來對環境有一個全盤性的了解。環境基因體具有高多樣性以及高複雜度,最有名的例子是,Craig Venter從一桶海水中萃取細菌DNA,經過基因體分析後,發現含有150,000,000bp非重複性的鹼基對、1,800個genotype以及1,200,000個過去沒有被記錄過的基因,因此,要描繪出真實的完整的環境基因體特性,需要大量的資料量 (Venter et al., 2004)。為了研究高度複雜的環境基因體,近期發展出兩個學門Metagenomics與Metatranscriptomics;Metagenomics是在DNA的層級,來分析環境基因體;而Metatranscriptomics是以RNA的層級來分析,可以補強Metagenomics不足的部分,除了可以分析各種基因的表現量,還能夠偵測環境中的RNA病毒族群。
早期Metagenomics與Metatranscriptomics實驗流程中,必須將環境的基因體DNA或cDNA轉植到E. coli體內,再進行定序分析,其過程繁瑣而冗長,而且得到的資訊量相對有限 (Trings et al., 2005);現在metagenomics研究,可以跳過繁瑣的分子生物學實驗技術,只需要將環境中的DNA或cDNA,直接進行次代定序(next generation sequencing)。 隨著定序技術的發展,定序資料的輸出量也越來越高,也就能夠呈現基因體真實的樣貌,至2010年,一次NGS定序所能夠得到的資料量已經達到100Gb的等級,因此,面對高度複雜的環境基因體的研究,NGS是最有效率的工具。
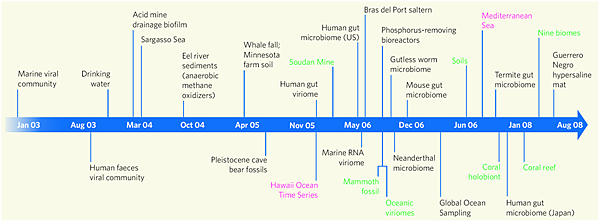
從2002年至08年間以有許多環境的基因體被解序分析,除了土壤和海水外,還包括腸內道微生物群的基因體,有越來越多證據顯示,腸內道的微生物群對動物的健康,扮演著重要的角色。





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
