目前發展成熟而被應用在許多研究的次世代定序技術中,被定序的library 大多為DNA而非RNA(定序原理請參照本部落格先前文章),在現有的mRNA-seq技術中,需先將RNA反轉錄為DNA,製備出DNA型態的library後,始能執行定序反應。針對mRNA-seq,近日Lira Mamanova團隊以Illumina定序平台為基礎,發展出可直接以RNA library做為定序材料的新技術,名為FRT-seq(Flowcell Reverse Transcription sequencing),此技術將RNA library注入flowcell內,並且直接在flowcell內執行反轉錄及bridge PCR,FRT-seq能完整保留RNA正負股的資訊(即strand-specific mRNA sequencing),除此之外,其製備library過程中不需經由PCR的放大,所以可以減少PCR所產生的錯誤(請參照本部落格PCR free文章)。
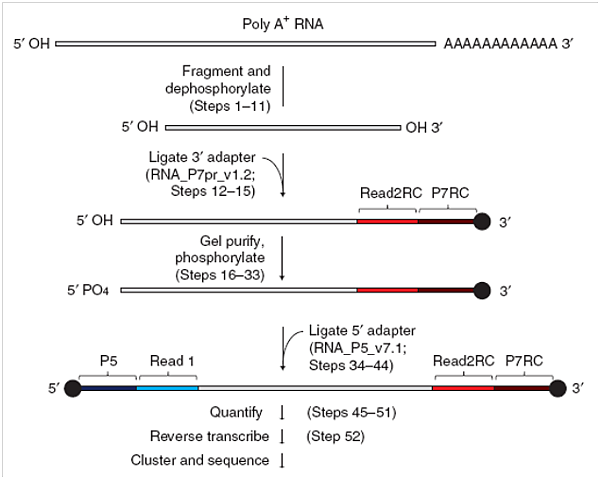
FRT-seq library的製備過程中,首先將mRNA純化出來並且打斷,再將RNA的3'及5'端分別接上兩段特殊設計且不同序列的adapter,經由qPCR及library分子大小的檢查後,便能以Illumina cBot將RNA library注入flowcell內,經由反轉錄及bridge PCR反應後,即可執行定序反應,流程如下圖所示。
在實驗的再現性上,如下圖所示,同一樣品經兩次FRT-seq實驗所定序出來的RPKM分析結果呈現Pearson correlation為0.993,代表此技術的實驗再現性是相當高的。

除此之外,FRT-seq完整保留RNA正負股的資訊,如下圖紅色所示,基因上同一區域正股及負股皆有被定序到的比例僅佔了6-7%的reads數,與目前已商品化試劑組製備的mRNA library相比較,若不經過特殊處理的mRNA library(關於能保留mRNA library正負股方向性的library製備技術,部落格後續會有文章介紹),如下圖藍色所示,其定序出來的結果無法顯示RNA的方向性。

FRT-seq具有library製備快速且能保留mRNA方向性的特性,雖然如此,此技術目前仍尚未被廣泛的應用,FRT-seq資料的正確性仍待更多研究資料的驗證。
參考資料:
1. Mamanova L, Turner DJ. Low-bias, strand-specific transcriptome Illumina sequencing by on-flowcell reverse transcription (FRT-seq). Nat Protoc. 2011 Oct 20;6(11):1736-47. doi: 10.1038/nprot.2011.399.
2. Mamanova L, Andrews RM, James KD, Sheridan EM, Ellis PD, Langford CF, Ost TW, Collins JE, Turner DJ. FRT-seq: amplification-free, strand-specific transcriptome sequencing. Nat Methods. 2010 Feb;7(2):130-2. Epub 2010 Jan 17.





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
