當我們想要研究一個物種,可是卻發現這個物種的序列資料 (DNA or RNA) 並不存在於世界上現有的資料庫。當我們遇到這種情況,便可以利用 NGS 的資料來進行所謂的 de novo assembly,藉此幫助我們獲取此物種的序列內容。而一個常見的 de novo assembly 流程概念如下:
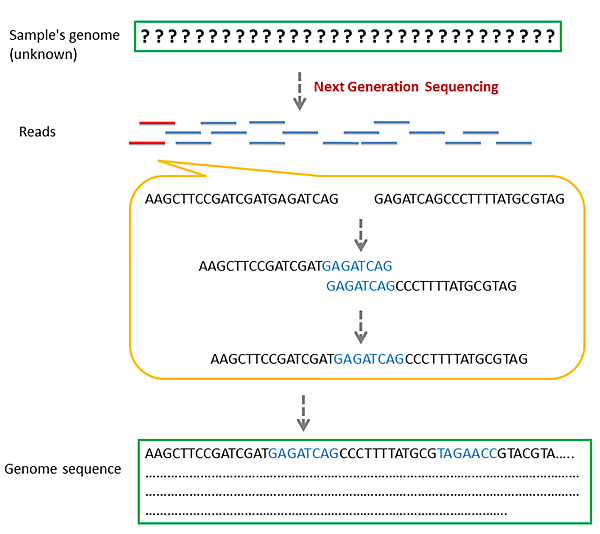
上圖表示將 NGS 的定序資料隨機抽取出兩條 reads (紅色線條),觀察此 reads 的尾端序列是否相同。如有某區域相同(藍色序列),則將此區域的序列合併連結起來,最後獲得一條更長的序列。透過這樣不斷的重複尋找,最終我們便可以將 reads 的資料還原回 genome 序列。
上述的過程是一個理想狀態,然而事實上有許多因素會造成我們組裝困難,我們將這些因素條列如後:
1.定序錯誤或核?酸多形性 (Sequencing error or nucleotide polymorphism)

從上圖可以看出,本來應該要合併在一起的 reads,卻因為定序錯誤或是核?酸的多型性造成尾端序列內容有兩處(紅色的字母 C 和 A )不同,因此程式無法判斷是否這兩條 reads 應該要合併在一起。
解決方法:
- 良好的樣品製備流程 (Good sample preparation)
- 選擇高品質的定序機器 (High quality sequencing machine)
- 定序深度加深 (Deeper sequencing)
- 序列組裝前先依據序列上的品質分數進行剪切 (Trim reads according to the quality scores)
2.定序偏差 (Sequencing bias)

從上圖可以看出,由於 GC content 的關係所造成的定序偏差,使得某些區域的序列不容易定出來(虛線部份),自然無法找出尾端序列一致的 reads
解決方法:
- 在樣品製備時使用特殊的藥劑甚至使用 PCR free 的方法 (Using special Kits or PCR free during sample preparation)
- 定序深度加深 (Deeper sequencing)
- 使用包含不同插入長度的對讀定序 (Different insert size of paired-end and mate-paired reads)
待續….





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
