GATK ( Genome Analysis Toolkit),一套用來分析DNA sequencing data,找尋genomic variation的工具,由Broad Institute開發。目前已被應用在幾個大計畫中,例如1000 genomes project、TCGA(The Cancer Genome Atlas)。而在今年七月也更新成第二版。
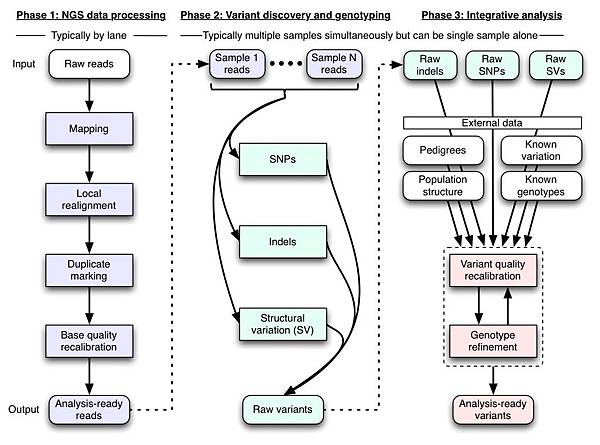
圖1. GATK流程圖
不同於類似的工具,GATK除了本身擁有相當多的walker供使用者使用外,更擁有一連串的校正步驟,目的就是為了減少false positive。舉個例子來說,GATK利用幾個public dataset (dbSNP、HapMap、Omni 2.5M chip),training出model來篩選掉一些false positive的SNP。
使用GATK的簡單分析流程如下:
- Alignment
- Remove PCR duplicates
- Local realignment
- Base quality recalibration
- Variant calling
- Variant quality recalibration
而在第二版修正了一些walker的使用方法(例如:base quality recalibration),此外還有新增了一些walkers:
- HaplotypeCaller-加入de novo assembly的概念去call variants,增加Sensitivity。
- ReduceReads-將沒有變異的consensus 區域merge起來,減少calling時間。
- Indel error model-第一版只有SNP的error model,在第二版加入了Indel的error model,使用2011年Ryan E. Mills發表在Genome Res上面的dataset。
References:
- McKenna A, et al., (2010) The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res, 20:1297-1303.
- McKenna A, et al., (2011) A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nat Genet, 43:491-498.
- http://www.broadinstitute.org/gatk/
全站熱搜





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
