在先前我們已經介紹使用於window7下使用bowtie(http://yourgene.pixnet.net/blog/post/92081187),今天在來介紹在window7下使用bowtie2的流程,bowtie2跟bowtie的最大差別在於bowtie2將reads比對到參考序列上時能允許indel的容錯率。
步驟一: 下載bowtie2
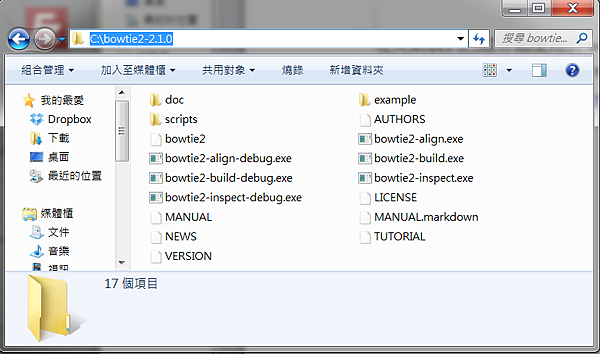
步驟二: 解壓縮及放到C槽下
並解壓縮到C:\下
步驟三: 使用命令提示員操作執行指令
bowtie2在操作方面與bowtie一樣需使用執行指令的方式來執行,在window7下則需開啟命令提示字元,快速開啟的方法是 1. 【開始】→2. 輸入cmd →3. Enter,之後請輸入cd c:\bowtie2-2.1.0,則命令提示字元的畫面會切換到前一步驟解壓縮的位置。

步驟四 :執行bowtie2,以paired-end read做alignment
在bowtie2的資料夾下,已經有將Enterobacteria phage lambda已知的基因體序列做完索引放在C:\bowtie2-2.1.0\example\index之下; 測試用的reads放在C:\bowtie2-2.1.0\example\reads下
最簡單的指令請輸入bowtie2-align -x example\index\lambda_virus -1 example\reads\reads_1.fq -2 example\reads\reads_2.fq -S lambda.sam

當輸入完上列指令後,螢幕會列出此alignment步驟的結果,共用10000個paired-end reads,其中有9166個paired-end reads是有unique的map到lambda上,42個paired-end reads是discordantly的map到lambda上,另有579條 single-end reads (broken reads) map到lambda上; 整體的alignment rate為94.97%
而打開資料夾可看到lambda.sam這個檔案

sam檔中紀錄了reads map到lambda的資訊,詳細資訊可參考(http://samtools.sourceforge.net/SAM1.pdf)
步驟五: 準備Import至IGV的檔案格式 (前置作業)
這個步驟需要使用到samtools,請先下載samtools ,並解壓縮至C槽。
於命令提示元輸入cd c:\samtools-0.1.12a_i386-win32
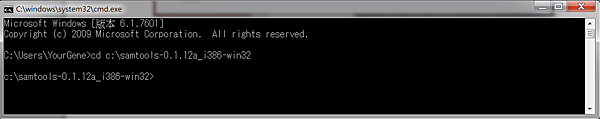
並依序完成下列3個指令
(1) samtools.exe view -bS c:\bowtie2-2.1.0\lambda.sam -o lambda.bam
(2) samtools sort lambda.bam lambda_sort
(3) samtools index lambda_sort.bam

完成後打開資料夾可看到有三個檔案產生

除了alignment的檔案之外,我們還需要lambda genome的註解資訊,請先到(http://www.ncbi.nlm.nih.gov/nuccore/9626243)下載lambda genome的genbank 格式另存到C:\samtools-0.1.12a_i386-win32下。

接著連到(http://www.ebi.ac.uk/Tools/sfc/readseq/), 並將lambda genome的genbank檔案上傳,並將INPUT FORMAT改成GenBank,OUTPUT FORMAT改成GFF並submit。

執行完的結果如下圖,請將檔案另存新檔為NC_001416.1.gff到C:\samtools-0.1.12a_i386-win32下

NC_001416.1.gff檔案打開內容如下

請將NC_001416取代成gi|9626243|ref|NC_001416.1|,並將第三行的” NC_001416 - source 1 48502…”移除,修改並存檔,結果如下

此時C:\samtools-0.1.12a_i386-win32的目錄有以下這些檔案

步驟六: 使用IGV來瀏覽reads map到lambda genome的情形
IGV的細節可參考(http://yourgene.pixnet.net/blog/post/96403107),使用上需先安裝JAVA (java載點) 及IGV (IGV載點)。
請先將IGV解壓縮到C槽下,並滑鼠點擊igv.jar開啟igv
開啟後的初始畫面如下

Import genome sequence (Genomes->Load Genome from File->選取C:\bowtie2-2.1.0\example\reference\lambda_virus.fa)

Import alignment file and annotation file (File-> Load from File-> C:\samtools-0.1.12a_i386-win32\ lambda_sort.bam及NC_001416.1.gff)

將alignment檔案及註解資訊輸入至IGV後的畫面如下

接者來看這次reads map到lambda genome的情形
例如在43,615-43,717的NinI基因上出現了一個SNV
reference的base為A,reads上出現的為C

接者看第二個例子,在6,712-6,814的E基因上,則有一個deletion

在這邊我們以bowtie2提供的範例檔來練習alignment, 轉換格式及IGV的使用,同樣的流程也可應用在有已知基因體序列的物種上。
如果需針對特定參考序列做alignment,需使用bowtie2-build這個指令來建立index,語法是bowtie2-build <input.fasta> <index檔名>
bowtie2的首頁也已提供了一些常用物種的基因體序列index檔

請連至(http://bowtie-bio.sourceforge.net/bowtie2/index.shtml)





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
