SSR(simple sequence repeat)是指兩個或多個核甘酸重複排列,重複的核甘酸稱為一個motif,重複的次數在不同的個體或族群中會有所不同,為一種多型性(polymorphism)的類型;以Jun在大豆芽的研究為例,如下圖一,研究團隊收集了不同區域的大豆芽(soybean aphid),分別在美國的俄亥俄州(OH)、伊利諾州(IL)、明尼蘇達州(MN)及加拿大的安大略省(ON),可發現不同區域的大豆芽,motif重複的次數會有所相異。

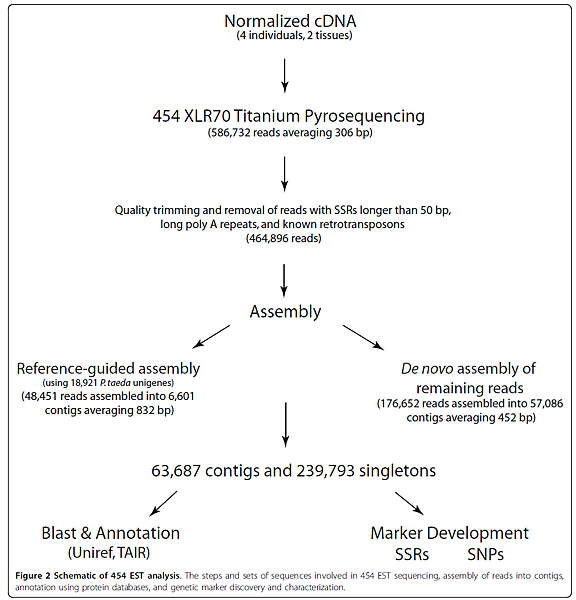
在Jun的研究中,為第一個針對大豆芽的genomic DNA使用NGS定序後,經過de novo assembly後的contigs從中找尋SSR marker及比較在不同區域中的genetic diversity。同樣的分析概念也可應用於RNA-Seq,在Parchman的研究中,定序了黑松(P. contorta)的RNA,透過de novo assembly及reference-guided assembly後的contigs,來找尋SSR marker,圖二為Parchman研究中的流程,可供想做相關研究的團隊一個參考。
References:
- TH Jun et al., Development of soybean aphid genomic SSR markers using next generation sequencing , Genome 54: 360–367 (2011) doi:10.1139/G11-002
- Parchman et al., Transcriptome sequencing in an ecologically important tree species: assembly, annotation, and marker discovery, BMC Genomics 2010, 11:180
全站熱搜





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
