A型流感病毒是人類最常感染的流行病毒 。以2009年發生大流行的H1N1為例,H1N1屬於A 型流感病毒,其病毒的基因是由人、鳥禽和豬的基因所組成。患有H1N1 的患者可能會有發燒、頭痛、全身性肌肉痠痛、咳嗽、喉嚨痛和鼻塞的症狀。而這種高度傳染的病毒有曾被世界衛生組織發出等級6的流行病警告。
由於病毒的高度抗原漂變(Antigenic drift )的能力,讓流感疫苗的效果有限。除此之外,病患若是同時感染兩種病毒,而使的不同病毒之間發生基因的片段重組(antigenic shift),使的在2009年發現了新型的病毒變種,而造成流感大流行。
Oseltamivir (奧司他偉,俗稱克流感)是其中一種針對H1N1的神經氨酸酶(Neurminidase, 簡稱NA,主要功能是幫助新生成的病毒脫離被感染的宿主細胞)有效抑制活性的藥物。而A 型流感病毒具有Antigenic drift和antigenic shift的特性,在神經氨酸酶基因發生突變,所以在2007-2008年,雖然只發現少數的對克流感有抗藥性的 H1N1 病例,但而後發現之後的H1N1亞型流感有99.4% 幾乎都有抗藥性。
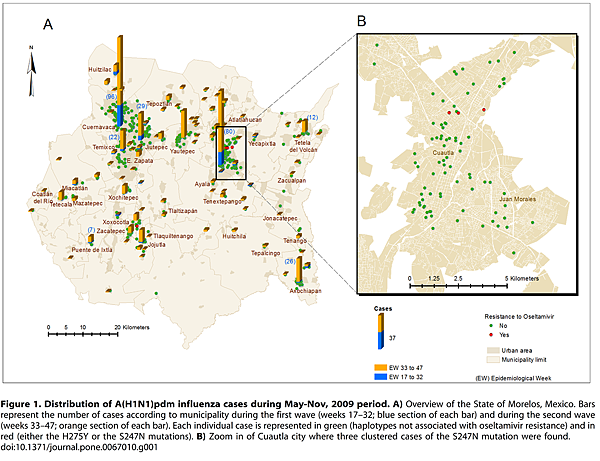
流感病毒具有高度的突變機率以及複雜的演化方式,所以NGS偵測病毒的多樣性時,必須以深度定序 (Deep sequencing) 才能達到好的偵測的量和值。但本篇作者想要以更簡易的方式以及更便宜的價格去建立可以快速監控當季流行病的模組,以利於方便監控病毒的演化以及可能有抗藥性的病毒株在某一地區的流行傾向。但要如何實行這個 low depth-large sample number approach 呢? H1N1 在墨西哥也曾經發生流行,本篇作者利用墨西哥Morelos州299個患有 H1N1 病患鼻子抹片檢體去製備NA基因的cDNA amplicon 作為本次實驗的材料。將檢體製備而成的amplicon,約5-7個同一時間或地點相近的以等量合併再一起,之後再一起製備library,依此類推的製備了48組不同 barcode 的 library。這樣大量 sample 合併的 library利用 NGS做淺度定序只能得到42,599條可用的reads,相對於每個 amplicon 只有144條 reads。但是這樣對於分析各個 amplicon 已經是十分足夠了。

這樣分析出來的結果發在299個檢體中發現了27 種H1N1病毒的亞型。將這些在核糖核酸層級就發生變化的病毒亞型 (quasi-species) 分別以數字標記。 其中有17個是胺基酸沒有改變的 (synonymous) 另外10個是胺基酸有變化的 (non-synonymous) 。以突變的位置去分類的位置約劃分為四組,如上圖的樹狀圖所示。可以發現最佔優勢的是IV組。

在 IV組大多是N248D的變形,多半與抗藥性無關。但其中有兩種病毒亞型是有關於抗藥性的突變,就是S247N和H275Y的病毒亞型。本篇作者還利用Sanger sequencing 來做檢驗結果的參考。
在這兩種定序技術的搭配之下,本篇作者發現以NGS 執行 low depth-large sample number approach 確實可以精確的檢驗出不同的病毒亞型。相對於墨西哥的Morelos州這樣小地理範圍測試,未來也期望可以延續用在大範圍的地理區檢測上,建立起一個快速且穩定的應用模式,來控制抗藥性病毒的蔓延。
Reference:
Juan TS, Mario HR, Rosa GB, Humberto VT, Ana CH, Pablo CH, Rene SL, Erik CV, Celso R, Lourdes GG and Jesus MB. Using High-Throughput Sequencing to Leverage Surveillance of Genetic Diversity and Oseltamivir Resistance: A Pilot Study during the 2009 Influenza A(H1N1) Pandemic. PLoS One (2013) 8: e67010.





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
