作者:洪郁豪 /有勁生物科技
1948 年,Mandel與Métais首次在人體循環血液中發現含有經細胞代謝產生的游離 DNA (cell-free DNA/cfDNA),其在血漿中所占的含量通常約在1 ng/mL至 10 ng/mL之間。隨後在1977 年,癌症與cfDNA的關聯性研究雖被提出,但相關應用卻是在非侵入性產前檢測領域才有較多重大突破。到了1994年,癌症病人的cfDNA被檢測出腫瘤相關基因的突變型,這些被認為由腫瘤細胞代謝產生的cfDNA突變型,被稱為 ctDNA (circulating tumour DNA);其中KRAS基因為腫瘤相關基因中最常被提出探討的,在過去腫瘤研究上占有舉足輕重的地位。KRAS基因的功能與細胞分裂增生有關,可藉由開啟或關閉功能來調控細胞生長;突變的KRAS基因會打亂細胞正常生長而引發腫瘤。
由腫瘤釋出至血液中的ctDNA含量常因過低,而難以被偵測到;即便到現在,臨床上檢測血液ctDNA含量的技術仍不成熟。隨著次世代定序技術發展,雖然許多代表性的檢測方式如:CAPP-Seq已有很大的進步,但準確率仍未能達到需求;特別是癌症初期的檢測結果多不盡理想。此外,不同病人血液中的ctDNA/cfDNA含量 (0.1%~1) 也會影響檢測的靈敏度。
CRISPR/Cas系統,為部份原核生物特有的後天免疫系統,可消滅外來的質體或者噬菌體,在近代時常扮演基因工程中編輯基因的角色。韓國的研究團隊嘗試將這項技術應用在提高cfDNA中ctDNA的比例,將其命名為CUT-PCR (CRISPR-mediated, Ultrasensitivedetection of Target DNA- Polymerase chain reaction);並參考 COSMIC (Catalogue of Somatic Mutations in Cancer) 資料庫上被登錄與癌症相關基因的突變點位置,尋找基因附近可供辨識的PAM序列,再透過特殊設計的gRNA (guide RNA),針對Wild-type(WT;即未突變的癌症基因)的專一性辨識後,發動核酸內切酶的攻擊(圖一上)。為了測試這項技術的可行性,研究團隊首先針對 KRAS基因序列設計人工質體並進行測試(圖一下)。方法是利用突變位點的錯位,讓代表突變型(Mutant)的各組質體發生突變,失去提供專一性辨識的PAM序列,之後用NcoI將這些質體剪切成線狀,再以CRISPR核酸酶進行處理。從圖二可觀察到,只有KRAS (WT)及Mutant 35G>A會被CRISPR核酸酶降解,其餘各突變組大致上不受影響。
圖一、CUT-PCR 作用原理以及其參考資料庫後的實驗設計
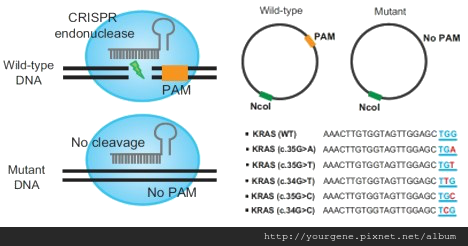
圖片來源:H Lee, S & Yu, J & Hwang, G-H & Kim, S & S Kim, H & Ye, S & Kim, K & Park, J & Y Park, D & Cho, Y-K & Kim, J-S & Bae, S. (2017). CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene. . 10.1038/onc.2017.281.
圖二、使用人工質體測試 WT 以及 PAM mutant 的核酸降解效果
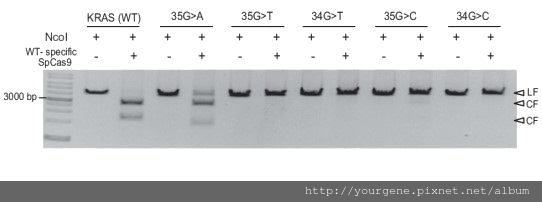
圖片來源:H Lee, S & Yu, J & Hwang, G-H & Kim, S & S Kim, H & Ye, S & Kim, K & Park, J & Y Park, D & Cho, Y-K & Kim, J-S & Bae, S. (2017). CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene advanced online publication 28 August 2017; doi:10.1038/onc.2017.281.
為了進一步確認CUT-PCR的效用,研究團隊利用Illumina targeted deep sequencing進行了類似實驗:模擬人體cfDNA狀況將WT質體與突變型質體以不同比例混合,然後做CRISPR核酸酶處理,之後再設計KRAS引子對,進行PCR製備library,然後定序。
從圖三左可觀察到,相較於未經處理的控制組(Control),經CUT-PCR法處理過後,混合質體中KRAS c.35G>T突變型序列的含量比例大幅增加。例如:原本含量僅佔0.01%比例的ctDNA突變型質體,在CUT-PCR處理之後,突變型序列含量顯著提升,幅度約達控制組的6倍。圖三右的四個實驗組(突變型質體含量各佔0.1%),經CUT-PCR處理後,混合質體中突變型序列含量也都有顯著提升(29.6到76.3倍)。
圖三、以不同比例混合 WT 與 Mutant 人工質體比較處理 CUT-PCR 後的差異
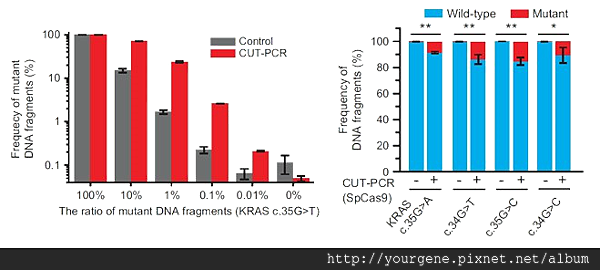
圖說:左圖是將KRAS(WT)與KRAS c.35G>T位點錯位的突變型質體,以100%、10%、......、0.01%、0%等不同比例混合後,比較有無CUT-PCR處理的結果。右圖是將KRAS(WT)各與四種ctDNA突變型質體─KRAS c.35G>A、KRAS c.34G>T、KRAS c.35G>C、KRAS c.34G>C混合(0.1% 比例)後,比較各組有無CUT-PCR處理的結果。 (圖片來源:H Lee, S & Yu, J & Hwang, G-H & Kim, S & S Kim, H & Ye, S & Kim, K & Park, J & Y Park, D & Cho, Y-K & Kim, J-S & Bae, S. (2017). CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene advanced online publication 28 August 2017; doi:10.1038/onc.2017.281.)
最後,研究團隊比較了8位不同階段的大腸癌病患與4位健康民眾的差異:將抽取自人體的cfDNA直接用上述CUT-PCR做處理,以針對Wild-Type KRAS基因為降解目標的sgRNA處理後,同樣也使用targeted deep sequencing來比較mutant與WT比例間的變化。從圖四a可觀察到隨著CUT-PCR處理次數的增加,ctDNA上升比例在各個突變型中越來越明顯,尤其在2、3、4、5、7 號病人與健康民眾上,有提升164至640倍效果的顯著差異。這項數據顯示,即便在第一期癌症病患的cfDNA中,經過有效處理後,也能夠忠實反映且成功偵測到ctDNA的訊號;而未罹癌的一般民眾也不容易發生偽陽性結果,有其珍貴的應用價值。
本實驗僅針對 KRAS基因,並由於突變位點能應用在 CRISPR/Cas系統辨識的PAM序列,所以才能成功利用。假以時日若能發現更多種類的CRISPR核酸內切酶,或者利用基因工程技術讓 CRISPR/Cas 系統辨識的 PAM 專一性有更多選擇的話,相信更能大大提高這項技術的實用性。
圖四、比較健康與罹癌 cfDNA 處理不同次數 CUT-PCR 後的差異
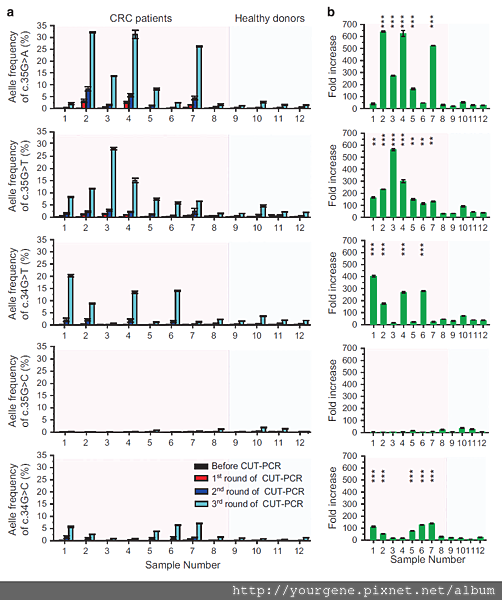
圖片來源:H Lee, S & Yu, J & Hwang, G-H & Kim, S & S Kim, H & Ye, S & Kim, K & Park, J & Y Park, D & Cho, Y-K & Kim, J-S & Bae, S. (2017). CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene advanced online publication 28 August 2017; doi:10.1038/onc.2017.281.
參考文獻
1. H Lee, S & Yu, J & Hwang, G-H & Kim, S & S Kim, H & Ye, S & Kim, K & Park, J & Y Park, D & Cho, Y-K & Kim, J-S & Bae, S. (2017). CUT-PCR: CRISPR-mediated, ultrasensitive detection of target DNA using PCR. Oncogene advance online publication 28 August 2017; doi: 10.1038/onc.2017.281
2. Wan JC, Massie C, Garcia-Corbacho J, et al.: Liquid biopsies come of age: Towards implementation of circulating tumor DNA. Nat Rev Cancer 2017;17:223–238.





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
