作者:洪郁豪/有勁基因
隨著次世代定序(NGS)技術的進步,近年來,基因定序的速度和輸出資料量大大提高。十年前的Genome Analyzer每次能產生的資料量最多只有1 Gb;如今,利用相同核心技術所建構的NovaSeq™6000,兩天內就能產出高達2 Tb的資料量。為了應付這麼大的資料產出量,就得將大量基因庫(library)放進每一輪的定序作業中同時讀取,這項技術稱為「多工處理(multiplexing)」。進入多工處理的所有基因庫在製備時,每條DNA片段上都會先被加入作為標籤(index)用的專一性基因序列,以便能在定序晶片內的單一通道(lane)中塞進大量的混合基因庫去同時進行定序。然而如此一來,經過多工處理後的基因庫定序資料也等於多了一層複雜性,所以在進入後端軟體分析之前,還得先通過一道「多工解訊(demultiplexing)」的步驟,將讀取到的資料先辨識並分門別類好才行。然而,Illumina次世代定序當初在開發基因庫多工處理技術時,曾遭遇標籤錯配(index misassignment)問題的嚴重挑戰;有鑑於此,便在官方白皮書上特別針對這個問題做了詳盡的介紹和因應說明。
近年來,Illumina為配合次世代新型定序晶片Patterned Flow Cells的應用,推出了Exclusion Amplification Chemistry(ExAmp Chemistry)酵素系統,主打優勢為:能節省成本、提高資料量的輸出、並減少定序所花的時間。然而相較於傳統Bridge PCR (橋式聚合酶鏈鎖反應)系統,ExAmp Chemistry酵素系統搭配Patterned Flow Cells晶片的這個方法卻反而造成更多的標籤錯配現象;對此,Illumina公司在其官方白皮書中指出,原因是大量基因庫混合上機的多工處理過程中發生了「標籤跳躍(index hopping)」的狀況。標籤跳躍所造成的這些錯位結果,讓接下來多工解訊階段的序列辨識和分類以及後續定序資料的分析都跟著出錯(詳見圖一)。
圖一、總覽基因庫混合上機時正常的多工處理(multiplexing)結果、以及發生「標籤跳躍(index hopping)的錯位結果
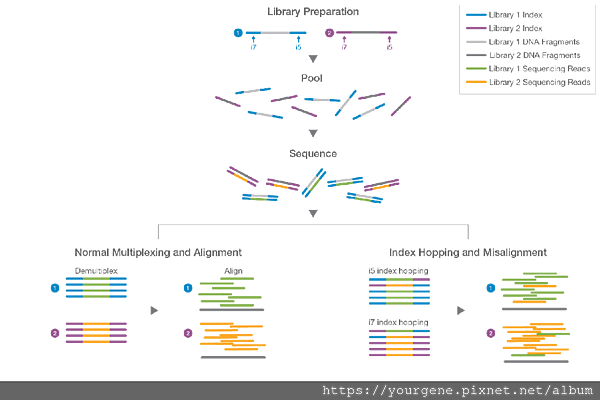
製備基因庫時,在每條DNA片段加上具專一性基因序列的i7和i5轉接子作為標籤,就可藉此附著到定序晶片(例如Patterned Flow Cells)上,讓大量的混合基因庫同時塞進單一通道(lane)內上機進行定序,這就是所謂的「多工處理(multiplexing)」技術。定序之後,再透過「多工解訊(demultiplexing)」過程,按照讀數將基因庫進行分類。然而,如果在「多工處理」階段出現了標籤跳躍的狀況,就會造成定序資料的錯誤分配,導致後端資料分析接連出錯。(圖片來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
那麼,標籤跳躍為何會發生?發生比例有多少?對定序過程和結果會帶來多大影響?該如何因應?以下就來分別摘要一下Illumina官方白皮書的說法:
1. 轉接子或引子對的殘留汙染會導致標籤跳躍的發生
基因庫製備過程中,轉接子(adapter)會被連接至目標基因片段然後附著到定序晶片上,之後再加入引子對(primers)去進行基因庫的增幅;基因庫製備好後,再利用切膠純化、或用能吸附核酸的磁珠去進行純化,將殘留游離的轉接子和引子去除掉。這個清除步驟若是做得不夠徹底,上機定序時就有可能發生標籤跳躍的狀況。下圖二「游離轉接子與標籤跳躍發生比例」的實驗結果顯示,上機定序的基因庫中若完全不含游離的轉接子或引子,標籤跳躍發生的比例為1.5%(藍線標示);在依序加入0 到35 nM不等份量的轉接子後(綠線標示),標籤跳躍發生的比例則從1.5%起跳,之後便依序增加(紫線標示)。
圖二、上機定序的基因庫若含有游離的轉接子,標籤跳躍的發生比例就會升高
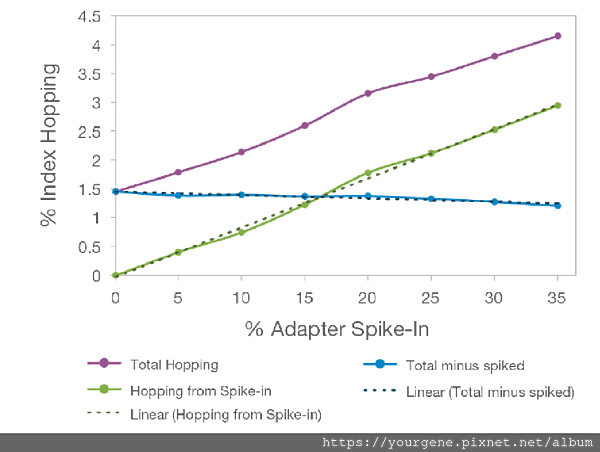
由圖可知,「標籤跳躍的發生比例」與「上機定序的基因庫中游離轉接子含量」之間具有正相關的線性對應關係。(圖片來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
2. 標籤跳躍程度的評估
利用含專一性基因序列的i7和i5轉接子在目標序列的兩端各標上標籤,然後將帶有雙重標籤的這些基因庫合併上機定序,接著進入多工解訊流程,之後便可對標籤跳躍的程度進行定量。下圖三是帶有不同雙重標籤組合的基因庫混合上機後所觀察到的標籤跳躍情況。綠色框框內的4支基因庫(702-502、704-505、705-503、706-506) 顯示為「有效的標籤組合」,其比例數值都在22.91~26.20%範圍之內。12個紅色框框內的基因庫為受標籤跳躍影響的「無效標籤組合」,其比例值在0.12~0.17%之間。從圖三這12個無效標籤組合的比例值來看,標籤跳躍的影響並沒有序列上的優先順序偏好。
圖三、從標籤污染比例評估標籤跳躍的程度
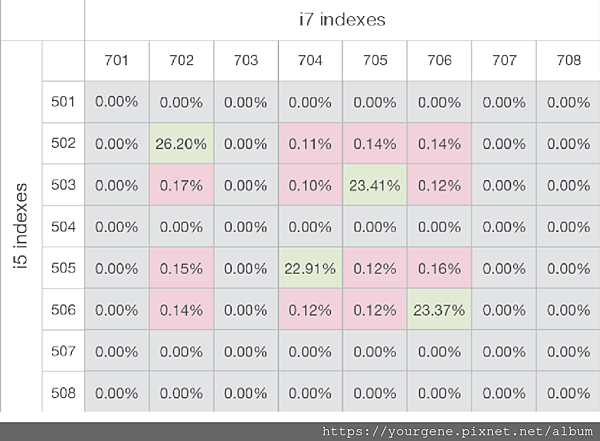
百分比標示各種雙重標籤組合出現的比例。矩陣中以標籤組合比例值為「有效」(綠色框)或「無效」(紅色框)來評估各基因庫發生標籤跳躍的程度。灰色框是未採用到的基因庫配對混合,當作對照組。從12個紅色框的無效標籤組合比例值來看,標籤跳躍的影響並沒有序列上的優先順序偏好。
(圖片來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
3. 標籤跳躍的影響
基因庫的製備方式也和標籤跳躍會不會出現有關。一般來說,轉接子黏合步驟之後不進行PCR增幅的基因庫製備方式,最容易出現標籤跳躍的狀況。舉例來說,相較於使用TruSeq™ Nano DNA Library Prep Kit試劑並且加入 PCR增幅步驟的製備方式,使用TruSeq™DNA PCR-Free Library Prep Kit試劑不進行 PCR增幅的製備方式,後者出現標籤跳躍的機會明顯升高(如圖四所示)。此外像是進行定序前「叢集生成(cluster generation)」的處理步驟時,相較於傳統Bridge PCR搭配Non-Patterned Flow Cells定序晶片進行增幅的方法,使用ExAmp Chemistry酵素系統搭配新型晶片Patterned Flow Cells的方法反而出現更嚴重的標籤跳躍情況;而且後者不論其基因庫如何製備,結果都是這樣(詳見圖四)。
圖四、比較不同定序系統的標籤跳躍差異
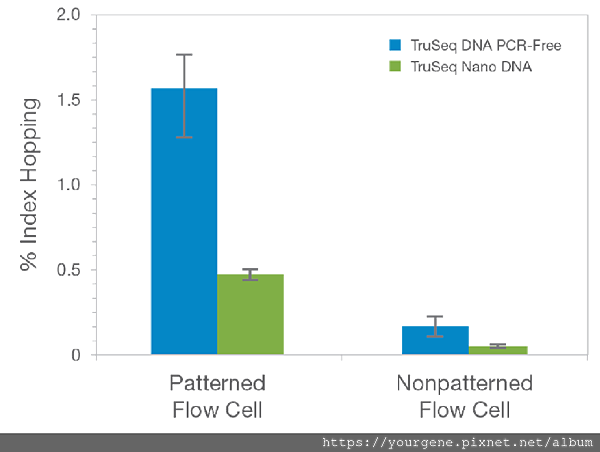
本圖比較TruSeq DNA PCR-Free(藍色標示)和TruSeq Nano DNA(綠色標示)兩種不同基因庫製備方式,搭配新型 Patterned Flow Cells與傳統Non-Patterned Flow Cells兩種定序晶片(X軸)所觀察到的標籤跳躍比例(Y軸)。結果發現,不進行 PCR 增幅的基因庫製備方式(TruSeq DNA PCR-Free),其出現標籤跳躍的機會明顯升高。而不論基因庫如何製備,使用ExAmp Chemistry酵素系統搭配新型晶片Patterned Flow Cells,其標籤跳躍出現的情況比傳統Bridge PCR搭配傳統定序晶片更高。(圖片來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
4. 標籤跳躍對 RNA 定序實驗的影響
標籤跳躍對RNA定序資料的後續分析究竟會造成多大的影響,Illumina官方拿在某些器官組織內表現量很高的「組織特異性標誌基因備註」的RNA定序樣品實驗為例來進行檢視。這個實驗所觀察的標誌基因是某個轉錄體的基因,這個轉錄體基因會在肝臟中大量表現,但在大腦中則表現一般。實驗將肝臟、大腦組織樣本分別標上不同標籤製備成基因庫後混合上機,此時,如何混合上機將會影響最後呈現的定序結果,如圖五所示。結果發現,肝臟與大腦樣品混合上機後,原先僅在肝臟才有高表現的標誌基因,其資料竟出現在大腦的樣品定序結果中(紅色框框標示),表示定序過程出現了會影響結果的標籤跳躍情形。如果是將兩組大腦樣品混合上機,就不會出現這樣的定序結果。由此可知,在做轉錄體表現的定序研究時,建議應該避免將來源差異性高的組織樣品混合一起上機。
圖五、標籤跳躍對 RNA定序分析結果的影響
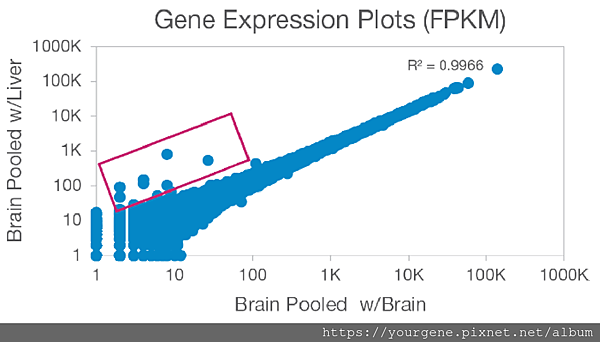
本圖以 FPKM表現圖(Fragments per kilobase million gene expression plots)比較肝臟和腦組織基因庫以不同混合方式上機定序的結果。Y軸是示肝腦組織基因庫一起混合上機定序的值,紅色框內標示在腦組織基因庫定序資料中出現肝組織才會有的高表現轉錄體基因資料(紅色框內的藍點),表示肝腦組織基因庫混合上機後,出現會影響結果的標籤跳躍情形。X軸是不同腦組織基因庫混合上機定序的值,這裡就沒有觀察到足以影響定序結果的標籤跳躍情形。 (圖片來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
5. 減少標籤跳躍的方法
Illumina公司在官方白皮書中,也將如何降低標籤跳躍發生機會的方法做了一個整理;如表一所示,主要可以從定序系統、基因庫的製備流程、儲存條件等方向來下手。例如:基因庫平時應該要貯存在-20℃ (不要貯存在4℃)的環境下。基因庫一經混合,就得盡快上機定序,如果未能及時上機,就得放回-20℃存放,且不得放超過一個禮拜。
Illumina 將雙標籤系統改良後,這幾年又推出了新式的雙標籤系統,所有96個樣品定序格的標籤序列均是獨一無二、沒有重覆的,因此即使發生了標籤跳躍,無效的標籤組合在後面定序分析時也能很容易就排除掉。此外,他們試劑組所提供用來阻隔游離轉接子用的酵素試劑也有進步;這個酵素試劑可以在基因庫製備過程中轉接子黏合之後,將游離轉接子的3ʹ 端預先做阻隔,以免後續上機進行Bridge PCR 時游離轉接子與錯誤的基因序列進行黏合,如此便可減少標籤跳躍發生的機會。關於Illumina新式雙標籤系統的詳細介紹,大家可以上他們的官網自行參考。
表一、減少標籤跳躍的最佳做法

本表由上到下依序羅列了5項減少標籤跳躍的建議做法。(1)採用Illumina新型的雙標籤系統來製備基因庫(標籤跳躍造成的無效標籤組合,會因無法分類而自動被系統排除掉)。(2)避免基因庫混合上機。(3) 將游離轉接子與引子對徹底清除乾淨。(4)未能即時上機的基因庫要依照建議保存在 -20℃的環境。(5) RNA 基因庫若要混合上機,要挑選來源組織差異不大的RNA 基因庫(避免基因表現高低差異大的基因庫彼此污染造成干擾)。(表格來源:Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746.)
備註:某基因在某器官組織中的表現量若總是比在其他器官組織中來得特別高或低,表示該基因在這個器官組織上有特異性的表現,因此可用來作為該器官組織的「組織特異性標誌基因tissue-specific marker (tissue-specific marker)」。
參考文獻
1. Illumina, Inc. (2018). Effects of Index Misassignment on Multiplexing and Downstream Analysis. Pub. No. 770-2017-004-D QB 5746. Retrieved from https://www.illumina.com/content/dam/illumina-marketing/documents/products/whitepapers/index-hopping-white-paper-770-2017-004.pdf





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
