作者:吳上豪/有勁基因
Illumina®公司在2015年推出HiSeq 3000/4000平台,並設計了新的文庫定序晶片(Patterned flowcell)以及新的酵素系統(Exclusion amplification chemistry)。新的技術妥善規劃了晶片上供基因文庫(library)擴增成叢集的空間;雖然大幅增加單一晶片的定序資料量,卻也產生了一些原本舊平台不會發生的問題,就是定序結果中會出現較高比例的「重複讀取資料(duplicate reads)」。(延伸閱讀:有勁部落格《Illumina Patterned Flow Cell簡介》/《Illumina Pattern Flow Cell平台定序注意事項》/《Illumina平台上的「標籤跳躍」現象》/《Illumina各型螢光頻率定序方式介紹》)
什麼是duplicate reads?
定序時會希望每一條文庫讀出的序列都是獨一無二的,但由於樣品特性、文庫製備、定序平台特性之故,一條序列有時候會定序出兩條(或更多條)完全相同的序列,這種重複讀取的序列資料,會被稱為「一組duplicate reads」。
全基因體定序或外顯子定序組裝流程中,重複讀取序列會被移除,造成實際可用來分析的資料比預期的更少。此外,在RNA定序的後續分析中,duplicate reads也可能會影響基因表現量的計算。
Duplicate reads的成因
常見的情況大致有以下幾種1:
1、原始樣品本身的序列相似度高,例如PCR擴增產物和高表現量的RNA就常出現duplicate reads。
2、製備文庫過程中PCR步驟:一般基因文庫的建構過程,為了將產物擴增到足以上機的濃度,會需要進行PCR去放大樣品;由於放大後會產生完全相同的序列,上機取樣時難免就有機會出現duplicate reads的情形。一般來說,若能依照文庫製備試劑組(kit)指定的樣品量及PCR循環數(cycle)的設定進行製備,duplicate reads的比例通常不會超過10%。但若一開始的樣品加入量偏少,以致之後必須增加PCR cycle的話,duplicate reads出現的比例就可能會提高。
3、使用non-patterned flowcell的定序儀時,演算法對叢集(cluster)的判斷有誤的話,也會出現duplicate reads。
4、因Patterned flowcell的定序儀平台特性所造成的duplicate reads,下文將對此進行說明。
比較HiSeq2500及HiSeq4000平台出現duplicate reads的情況差異
QCFAIL網站2針對HiSeq2500及HiSeq4000平台上出現的duplicate reads做了一些比較,發現HiSeq4000平台得到的資料,其實有一大部分資訊都是duplicate reads(如圖一所示)2。
圖一、HiSeq 2500及 HiSeq 4000平台定序完成後的duplicate reads表現比較
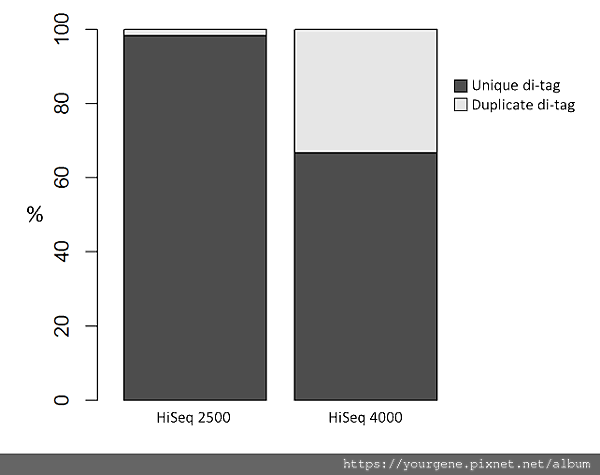
HiSeq 4000所得到的資料中有33%是duplicate reads,遠大於HiSeq 2500的情況(2%)。(圖片來源:Wingett, S. (2017 Mar 2). QCFail website by Babraham Bioinformatics & SciLifeLab.)
由於Illumina®平台會將定序晶片劃分成多個區域並分別拍攝照片,再將照片中各光點的資訊轉換成核苷酸序列。一張照片所記錄的區域稱為一個tile,定序資料會記錄每一條序列所在的tile編號並定位座標。Duplicate reads在晶片上的位置,也可以藉由這些資訊得知。QCFAIL網站的比較結果發現,整體來說,不管是在HiSeq2500或HiSeq4000平台,duplicate reads在晶片上的分布皆是隨機的,並不會特別集中在晶片上的某個區域。但若細看duplicate reads組內分布的相對位置,會發現HiSeq2500平台幾乎所有組別duplicate reads的重複序列資訊皆分布在不同的tile上,而HiSeq4000平台大部分duplicate reads組別的重複序列資訊則都出現在同一個tile上。這表示在Patterned flowcell平台上進行定序時,大多數duplicate reads的重複序列資訊都是在相近位置上產生的(見下圖二)2。
圖二、Duplicate reads在不同平台的分布情形
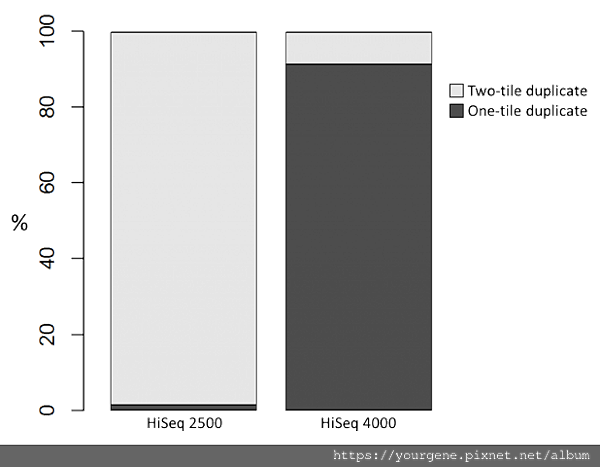
圖片來源:Wingett, S. (2017 Mar 2). QCFail website by Babraham Bioinformatics & SciLifeLab.
QCFAIL網站將上述HiSeq4000平台「出現在同一個tile上」的各組duplicate reads挑出,計算各組duplicate reads組內訊號的X軸、Y軸相對距離並作圖(如圖三所示)2,結果發現大部分duplicate reads的組內訊號皆相距不遠。有趣的是,若放大觀察座標差少於200像素的組別,會發現這些分布點所呈現出來的形狀並不像是隨機分布的,反而和patterned flowcell上的孔洞形狀很相似。
由以上結果可知,「HiSeq4000平台出現大量duplicate reads」的情況並非偶然發生,而是HiSeq4000平台或其定序原理所造成的特有現象。
圖三、HiSeq4000平台各組duplicate reads的組內分布間距
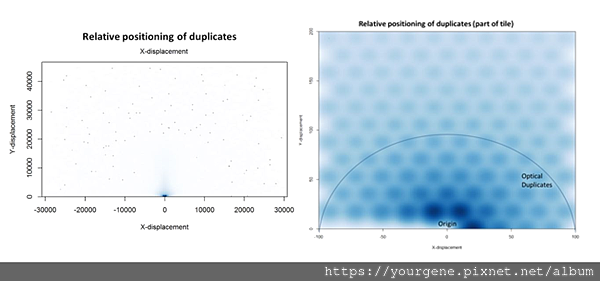
左圖是一整個tile中各組duplicate reads組內分布的相對距離。右圖僅取分布位置較相近的那些組別作圖。X、Y軸的數字代表duplicate reads在FASTQ檔案中的座標相減後的數值。(圖片來源:Wingett, S. (2017 Mar 2). QCFail website by Babraham Bioinformatics & SciLifeLab.)
HiSeq4000平台上出現duplicate reads的原因
根據以上的探討,HiSeq4000平台發生大量duplicate reads的原因有可能來自patterned flowcell的挖洞設計以及因應而生的序列判讀原理。傳統Non-patterned flowcell平台進行定序時,每一條序列在橋式聚合酶連鎖反應(bridge PCR)後所產生的叢集(cluster)其形狀、大小不同,因此在定序過程中,儀器必須定義每一個叢集的範圍才能進行圖片資訊分析。相較之下,Patterned flowcell平台已經規劃出各叢集的生長範圍;對儀器來說,從每一個孔洞中的叢集所得到的資訊即為一筆資訊。當基因文庫注入晶片時,難免殘留一些孔洞未被基因文庫佔據,這些空孔洞若在橋式聚合酶連鎖反應產生叢集的過程中,被鄰近孔洞中意外脫落的文庫流入並進行叢集擴增,就會造成兩個孔洞出現完全相同叢集的情形,定序時便會出現「出現兩筆完全相同的序列」,在後續分析時會因此被判定為duplicate reads。
如何減少duplicate reads的發生
既然duplicate reads是橋式聚合酶連鎖反應過程中,意外掉出的文庫佔據定序晶片的空孔洞進行叢集擴增所造成,那麼「增加注入晶片的文庫濃度」,讓定序晶片上所有孔洞在叢集擴增之前都能夠盡量被佔滿,應該是最直接有效的解決方式。雖然增加文庫的上機濃度可以減少空孔洞及duplicate reads的發生,卻同時也會增加單一孔洞出現多個叢集的發生率,造成上機品質與可用定序量的下降(如圖四所示)3。
由此可見,不同機台或不同類型的文庫,其最佳上機濃度皆不相同;因此尋找最佳上機濃度對於patterned flowcell平台來說,仍是一門重要的課題。
圖四、文庫上機濃度對上機結果的影響
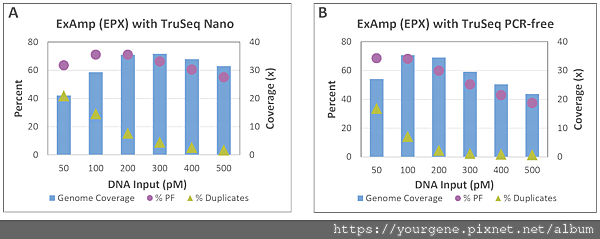
A: 一般DNA文庫;B: PCR-free文庫。X軸是基因文庫的上機濃度。紫色圓圈是晶片孔洞中含單一文庫叢集佔所有孔洞的比例,其數值對應在左側的Y軸。綠色三角形是duplicate reads所佔的比例,數值對應在左側Y軸,數值越高表示duplicate reads發生的情況越嚴重。藍色柱體是定序資料移除duplicate reads及品質不良訊號後的序列覆蓋倍率,數值對應在右側的Y軸,數值越高表示可有效分析的數據越多。(圖片來源:Illumina®. (2016). Optimizing Cluster Density on Illumina Sequencing Systems.)
參考文獻
1. Hadfield, J. (2016 May 23). Increased read duplication on patterned flowcells- understanding the impact of Exclusion Amplification. Retrieved from Enseqlopedia.com. http://enseqlopedia.com/2016/05/increased-read-duplication-on-patterned-flowcells-understanding-the-impact-of-exclusion-amplification/
2. Wingett, S. (2017 Mar 2). Illumina Patterned Flow Cells Generate Duplicated Sequences. Babraham Bioinformatics & SciLifeLab. Retrieved from QCFail.com. https://sequencing.qcfail.com/articles/illumina-patterned-flow-cells-generate-duplicated-sequences/
3. Illumina®. (2016). Optimizing Cluster Density on Illumina Sequencing Systems. Retrieved from Illumina, Inc. https://www.illumina.com/content/dam/illumina-marketing/documents/products/other/miseq-overclustering-primer-770-2014-038.pdf





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
