隨著定序價格的降低,以及Illumina定序長度的增加,越來越多人改用Illumina HiSeq或MiSeq平台進行16S metagenomics sequencing。定序所需之library製備方式,也不斷地在改進。以下列出幾種常見的幾種16S metagenomics sequencing之library製備方式,以及各自的優點與缺點。
採用 Barcoded 16S rRNA primer & Illumina TruSeq DNA Library Preparation
首先我們須先根據待定序的樣品數目, 設計不同的 16S-barcoded-primer。以16S的V1-V2 region為例,在27F以及338R primer 的5’端加上6-mer的barcod序列 (如下表,粗體底線的部分是barcode序列,其餘則是原本的16S primer序列)。
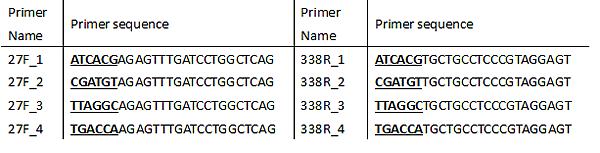
使用上述primer 序列放大樣品中的16S region之後,再將各樣品等量混和成一管,並根據Illumina TruSeq DNA PCR-Free Library Preparation製備library,流程圖如下:
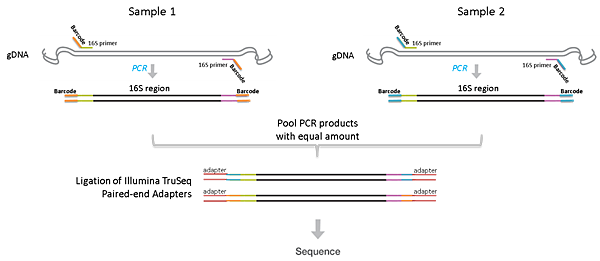
此方法之優點為:將多個樣品pool成一管,製備一個library,可減少library製備之成本;缺點為:由於將多個樣品混和在一起,容易在後續PCR amplify的過程中產生Chimera reads,可選用 PCR-Free之library製備方式,以避免Chimera reads的產生。
採用 Single-indexing 16S rRNA targeting Library Preparation
Caporaso等人在2011年提出的方法,我們可以設計長片段之PCR primer,包含Illumina adapter、index以及16S primer,再利用此 long overhang PCR primer去放大樣品中的16S rRNA 區域,如下圖所示:

此方法設計的長片段 primer中,除了16S region的primer以及Illumina adapter之外,還另外加上了”Pad”和”Linker”的序列,這兩段序列是為了避免長片段之primer容易形成dimer而添加的。此優點之方法為:相較於使用Ligation添加Illumina adapter,使用PCR的方式,library製備的效率較好,且PCR完後就可以直接上機定序,減少library製備的中間步驟;缺點為:此方法之每一樣品均需要設計不同的primer set,primer合成價格較高,另外長片段primer PCR,也容易造成primer-dimer、hairpins、專一性降低等問題。
採用2-step PCR & duel-indexing 16S rRNA targeting Library Preparation
此方法為Illumina官方網站上面目前提供的16S rRNA library preparation protocol。相較於Single-indexing 16SrRNA library preparation,只有在3’端的adapter上放置index,Duel-indexing 在5’端以及3’端的adapter都有放置index。另外使用兩階段的PCR,第一組PCR primer包含部分Illumina adapter序列以及16S primer序列,第二組PCR primer則包含完整的Illumina adapter序列以及index部分。詳細圖示如下:
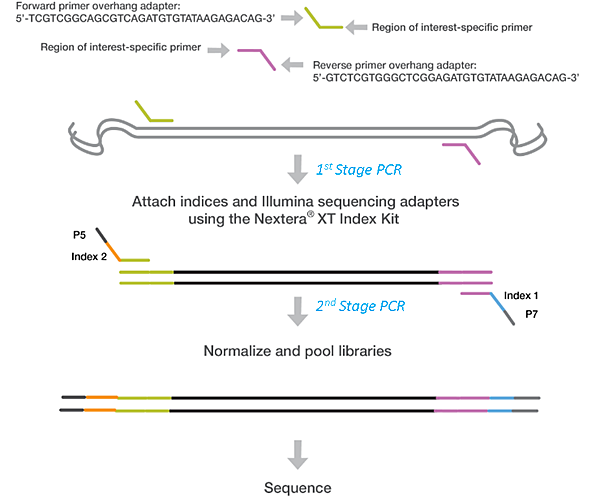
此方法之優點為:使用PCR方式加上Illumina adapter序列,library製備之效率較高,且使用二階段PCR,第一組PCR primer所有的sample pool都可以共用,且可以避免長片段primer在PCR過程中的專一性問題。缺點則為:雖然第一組primer可以共用,但第二組primer(含index)仍然需要每個樣品各自設計,故primer設計成本以及library validation的價格較第一種方法高。
上述三種16S rRNA library製備方式,研究者可以根據自己的需求,成本考量,而選擇不同的製備方式,目前這三種方式都可以找到許多paper採用,其中第二種方式,是目前Earth microbiome project上所提供的16S rRNA Amplification Protocol,且每個protocol中的16S primer,也可根據研究者想要看的不同區間來做替換,增加實驗的彈性。
參考文獻:
- Lazarevic, Vladimir, et al. “Metagenomic study of the oral microbiota by Illumina high-throughput sequencing” J Microbiol Methods (2009) 79(3): 266–271.
- Caporaso, J. Gregory, et al. “Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample” PNAS (2011) 108(Suppl 1): 4516–4522.
- Caporaso, J. Gregory, et al. “Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms” ISME J. (2012) 6(8): 1621–1624.
- Illumina: 16S Metagenomic Sequencing Library Preparation.




 留言列表
留言列表 {{ article.title }}
{{ article.title }}
