作者:黃存操/有勁基因
「精準醫療」和一般醫療不同之處,在於精準醫療會根據患者的致病基因變異,來決定該施予病人何種治療方式。因此,精準的基因檢測是實施精準治療的必要前提。
人類基因有兩萬多個,每個都是由一長串核苷酸序列所組成;基因中每個位點的核苷酸都有可能因環境刺激或隨機因素而被改變,這些改變統稱為「基因變異」。傳統的桑格(Sanger)基因定序法適用於單一基因有限長度區段內特定位點的變異檢測上。其他定序方法,例如目標基因檢測⼀BRCA1/2基因檢測,就可以針對BRCA1與BRCA2這兩個與乳癌/前列腺癌相關基因進行表現區域的變異檢測。倘若希望畢其功於一役,將人類樣品中兩萬多個基因表現區域的所有變異一次檢測完,就得用到全外顯子定序(Whole-Exome Sequencing;簡稱WES);這項檢測目前已經廣泛應用在多種臨床醫學研究上了。
全外顯子定序若要應用在臨床的治療評估上,就得先對全外顯子定序的檢測進行確效(validation),也就是要先確認該檢測是否能達到所要求的效果,以及可否得到足夠準確的檢測結果。進行確效時,須遵循試驗指南(guidelines)對設計驗證步驟的要求;經試驗後再將數據蒐集並量化為「準確度」以便判定效果。全外顯子定序的確效(WES validation)該如何進行,2016年美國Rennert, H團隊發表在《npj Genomic Medicine》期刊上一篇關於癌症精準治療的論文1有提供詳細的說明。本文將針對此論文完成WES確效的過程做個簡介。
紐約州衛生部在2016年有制定一個《次世代定序體細胞遺傳變異檢測指南NGS guidelines for somatic genetic variant detection》2,適用於所有類別體細胞遺傳變異的次世代定序檢測;Rennert團隊的全外顯子確效分析流程便是根據這個試驗指南設計出來的,詳見下圖一。
圖一、Rennert, H團隊的WES確效試驗分析流程圖

研究團隊的WES確效步驟為:(1)從人類組織切片取得樣品並(2)將癌細胞與正常細胞分離後,(3)使用HaloPlex基因檢測試劑分別為兩組細胞建立基因庫並確認品質,然後便(4)放入次世代定序儀進行基因定序,(5)定序參考序列採用的是hg19,(6)在取得BAM格式的定序資料檔案後,接著進行變異位點偵測的生物資訊分析,產出VCF檔格式的分析資料,之後再(7)利用IPM分析流程去對比多個變異資料庫及(8)取得確效評估的結果,然後(9)產出結果報告並(10)存成醫療紀錄。(圖片來源:Rennert, H. et al. npj Genomic Medicine. 2016, Jul; 1:16019-16029.)
整個確效試驗流程中所需的樣品個數與達標門檻皆須符合試驗指南的要求,才算確效完成。例如:Rennert, H研究團隊進行資料確效(WES accuracy, sensitivity and specificity)時使用了4個HapMap DNA樣品、2個Horizon FFPE樣品、以及57個能分出正常/癌化細胞的組織切片樣品來評估檢測結果的靈敏度與特異性,如下圖二所示。在評估突變的分析靈敏度(analytical sensitivity)時,則用了56個含有陽性突變的細胞株稀釋樣品、以及15個作為驗證用的腫瘤組織樣品。在精確度(Precision within run)與可重複性(Reproducibility between run)評估方面,也有相對應的樣本數目與判斷門檻。
圖二、基因檢測資料確效相關的樣品個數與判斷門檻
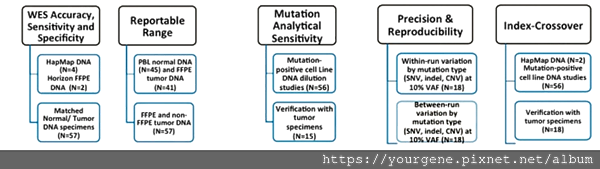
圖片來源:Rennert, H. et al. npj Genomic Medicine. 2016, Jul; 1:16019-16029.
流程最後(步驟9、10),將實驗結果依照試驗指南的要求列出並寫成確效報告。一個像此篇論文的全外顯子定序的試驗,從樣品取得到報告產生大約需要三週的時程。用於癌症精準治療的基因或變異位點分析資料中,以下兩項是最讓人感興趣、且可以被明確分析的:
- Clinically relevant genes:臨床上顯示與癌症有關聯的基因
- Clinically actionable mutations:臨床上已有對應治療方式的基因變異位點
在此要特別強調,全外顯子定序的檢測能力表現會因為以下因素而有所差異:樣本來自不同取樣方式或組織、源自相同組織的不相同基因、基因變異位於相同基因上的不同位點。Rennert團隊此篇論文的對參考資料其所有外顯子的檢測靈敏度(sensitivity)、特異性(specificity)以及陽性預測值(PPV),分別為95.7%、99.9%和99.2%。在前瞻性分析(prospective analysis)方面,有8成2的案例可以檢測出臨床上有治療辦法的基因變異位點或與癌症有關的新變異。
全外顯子定序的確效之所以會這麼複雜,是因為它一次可偵測高達兩萬多個基因,約計三千萬個位點,與一般生化檢測的數十到數百種項目相較高出10萬倍。同理可知,要想判別全外顯子定序的檢測是否有效,就得考量許多實驗上的變因,以致全外顯子定序的確效過程會變得如此複雜;即便想去比較不同全外顯子定序方法之間的優劣,也都不是件容易的事呢。
備註
紐約州衛生部《次世代定序體細胞遺傳變異檢測指南NGS guidelines for somatic genetic variant detection》,主要內容包括以下幾項:
● 標準作業流程(SOP; Standard Operation Procedure):實驗流程敘述必須一步步加以說明,且確效的步驟必須明示門檻及提供指示。
● 品質管制(QC; Quality Control):包含對實驗品質與後續數據分析的品質要求。
● 控制組(Control Group;又稱對照組):必須包含NTC控制組(No Template Control) 、指定的陰性控制組(Negative Control)、每個run都要有陽性控制組(Positive Control)。
● 報告(Report):列出檢測報告中必須提供的資訊,詳細規定請參照原文。
● 確效(Validation):除了說明確效必須要做的事情外,也規定最少需有3個確定的參考樣品來測定實驗室的錯誤率(laboratory specific error rate)、此錯誤率需要小於2%。對於初始確效(Initial Validation)最少需要有50個病人檢體;完整確效(Full Validation)中,對於每種型態的變異則最少需要10個已被定序且確認的陽性樣品。不同的變異例如說單核苷酸變異(SNV), 插入缺失(Indels), 拷貝數變異(CNVs)等等的要求也不同,在指南內有詳細說明。
○ 精確度(Precision within run):對於那些其存在的比例低到接近偵測靈敏度極限的變異,最少得提供3個陽性樣品(有檢測出這些基因變異的樣品)的結果證明,且這些基因變異需在同一個run中經過3次重複操作的確認(每次操作皆授予不同的barcode標示條碼)。
○ 可重複性(Reproducibility between run):對於那些其存在的比例低到接近偵測靈敏度極限的變異,需要證明這個結果具有可重複性;於此,必須做3個不同runs的重複確認(每個run皆授予不同的barcode標示條碼)。可能的話,應由不同的技術人員在不同的日子去操作這項確效實驗。
○ 分析靈敏度(Analytical sensitivity):用細胞株去混合樣品,來量化分析靈敏度。這個項目需要使用3到5個病人的樣品來進行查證(verification)。
參考資料
1. Rennert, H. et al. Development and validation of a whole-exome sequencing test for simultaneous detection of point mutations, indels and copy-number alterations for precision cancer care. npj Genomic Medicine. 2016, Jul; 1:16019-16029. Retrieved from https://doi.org/10.1038/npjgenmed.2016.19
2. Cuomo, A.M. et al. (2016, Mar). "Next Generation" Sequencing (NGS) guidelines for somatic genetic variant detection. New York State: Department of Health. Retrieved from https://www.wadsworth.org/sites/default/files/WebDoc/1300145166/NextGenSeq_ONCO_Guidelines.pdf





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
