作者:邱翎雅/有勁基因
根據2018年WHO世界衛生組織的統計資料,全球平均每六人就有一人因癌症死亡1;癌症防治因此一直是全球衛生組織機構努力的方向之一。
基因突變是誘發癌症形成的主要原因;DNA若持續暴露在致癌因子下,突變就會隨著時間累積,在DNA中形成像分子指紋般特別的印記。不同類型的致癌與突變過程會形成不同的印記,也就是所謂的「突變特徵(mutational signatures)」。DNA突變特徵大部分是由外在的致癌因子所引起,例如:吸菸、紫外線照射或黃麴毒素等;有些則是和細胞內調控機制有關,例如:DNA 修復機制失常、DNA編輯酶突變、APOBEC胞嘧啶脫氨酶突變等等2,3。COSMIC(Catalogue of Somatic Mutations in Cancer)資料庫收錄很多和癌症有關的突變特徵;在2015年3月的第二版4中,COSMIC收錄了維康桑格研究所(WTSI; Wellcome Trust Sanger Institute)從40種癌症的10,952個全外顯子和1,048個全基因體序列中所拆解出的30種突變特徵(如圖一所示),其癌症資料來自美國癌症基因體圖譜計畫(TCGA; The Cancer Genome Atlas)和國際癌症基因體聯盟(ICGC; International Cancer Genome Consortium)5 。
圖一、COSMIC 2015年3月第二版所收錄的30種突變特徵
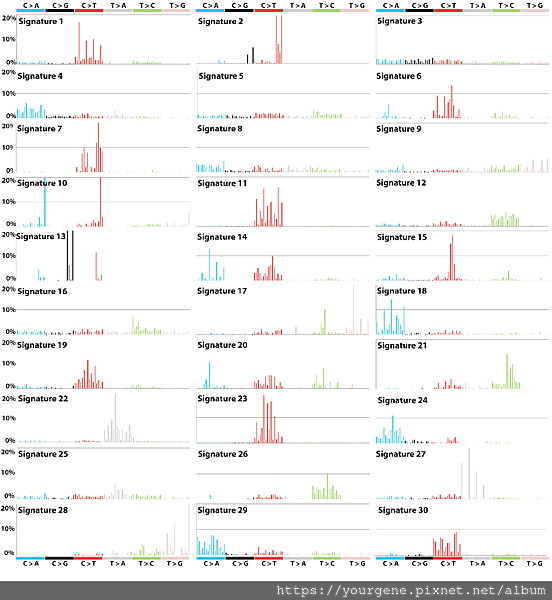
圖片來源:COSMIC. (2015 v2). Mutational Signatures (v2-March 2015)
突變特徵主要是用下面這6種不同的鹼基取代模式來表示:C>A、 C>G、 C>T、 T>A、 T>C和T>G註1。找到突變點後,再朝突變點的兩邊各往5端及3端囊括一個核甘酸,就是這個突變點的「三核甘酸組合(tri-nucleotide context)」,如圖二所示6 。換句話說,突變特徵共有96種三核甘酸組合的類型:4 (A/T/C/G)× 6 (C>A/C>G/ C>T/ T>A/ T>C/T>G) × 4 (A/T/C/G)= 96。圖三COSMIC所列出的突變特徵Signature 1,其C>T突變分布的比例就比較高。
圖二、三核甘酸組合示意圖
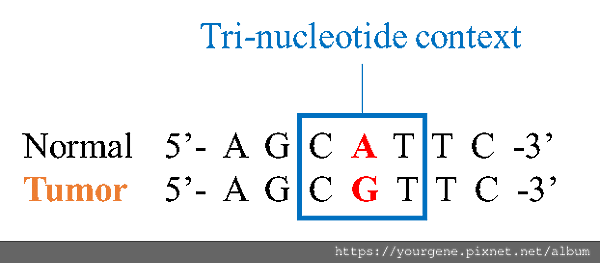
找出突變點A>G,並分別往突變點的前後(5端和3端)各囊括一個核甘酸C和T,組合成CA>GT三核甘酸組合。(圖片來源:邱翎雅/有勁基因)
圖三、 Signature 1
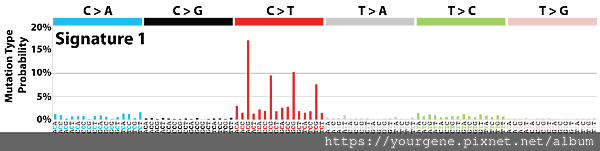
X 軸為96種三核甘酸可能組合,Y軸為某種突變類型(突變點的三核甘酸組合)所佔的比例。本圖突變特徵Signature 1是內在突變過程「自發性5′-甲基胞嘧啶脫氨作用」的結果,C>T的突變比例較高。(圖片來源:COSMIC. (2015 v2). Mutational Signatures (v2-March 2015))
從不同類型的突變特徵,我們可以對應到不同的致癌因子和癌症種類。例如圖三這個和年紀有關的突變特徵Signature 1就可以在所有癌症類型中找到,由於年紀越大,基因突變數量隨著時間累積越多,罹癌風險和機率也就越高。COSMIC的突變特徵Signature 7則是基於長期暴露於紫外光照射下所誘發的突變,可以在患有皮膚癌病人的身上觀察到2,3。由於人類的DNA會保有突變持續過程中所留下的印記,因此我們可以藉由癌細胞DNA的突變特徵去回溯誘發腫瘤的原因,有助癌症的預防與治療。目前雖然還有許多突變特徵不清楚,但相信未來會有更多更精確的突變特徵資訊被釐清。
至於突變特徵要如何利用非負矩陣分析法(Non-negative Matrix Factorization; NMF)從定序結果分析出來?以及2020年6月COSMIC突變特徵的最新版本v3.1內容,筆者之後再為大家詳細介紹。
註1:這時,大家可能會疑惑為什麼不是12種模式呢?原因是科學家根據胡斯坦鹼基對(Watson–Crick base pair),將G>T突變併在C>A突變類型,G>C併在C>G突變類型中,以此類推之故 。
參考文獻
1. World Health Organization. (2018, Sep. 12). Newsroom: Cancer. Retrieved from https://www.who.int/news-room/fact-sheets/detail/cancer
2. Alexandrov, L.B., & Stratton, M.R. Mutational signatures: the patterns of somatic mutations hidden in cancer genomes. Curr Opin Genet Dev. 2014 Feb;24:52-60. Retrieved from https://doi.org/10.1016/j.gde.2013.11.014
3. Helleday, T., Eshtad, S., & Nik-Zainal, S. Mechanisms underlying mutational signatures in human cancers. Nat Rev Genet. 2014 Jul;15(9):585-598. Retrieved from https://doi.org/10.1038/nrg3729
4. COSMIC. (2015). Mutational Signatures (v2-March 2015). Retrieved from https://cancer.sanger.ac.uk/cosmic/signatures_v2.tt
5. Alexandrov, L.B., et. al. Signatures of mutational processes in human cancer. Nature. 2013 Aug;500(7463):415-421. Retrieved from https://doi.org/10.1038/nature12477
6. Alexandrov, L.B., et. al. Deciphering signatures of mutational processes operative in human cancer. Cell Rep. 2013 Jan;3(1), 246-259. Retrieved from https://doi.org/10.1016/j.celrep.2012.12.008





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
