作者:林志鵬/有勁基因
場景:辦公室一隅
人物:部落格總編輯(以下簡稱「編」)、本文作者(以下簡稱「帥」)
編:……!!又是你!
帥:親愛的編輯早安,這是我這次的高品質稿件,認同請分享。
編:你每次文章都要挑戰我的極限,校稿都校到懷疑人生,還想要拿高分的評價?要high pass是不可能的!

圖片來源:GIPHY. You Shall Not Pass. Lord of the Rings gif. Retrieved from http://gph.is/1bgR8Yz
帥:可是我不要high pass,我這次要~~low pass!
編:!?
本文開始
=============================
利用NGS進行染色體DNA變異研究時,為了讓變異的偵測結果具備一定的可信度,我們通常會希望定序深度(sequencing depth;需要的定序讀數reads)至少要達30倍以上(一般會用英文「30X」表示)。如下圖一所示1,定序深度大約在30倍時,正確偵測的可能性就可以達到99.5%。
圖一、定序品質達到Q30的前後,「正確偵測異型合子出現單核苷酸多型性的機率」與「定序深度」之間的相關性
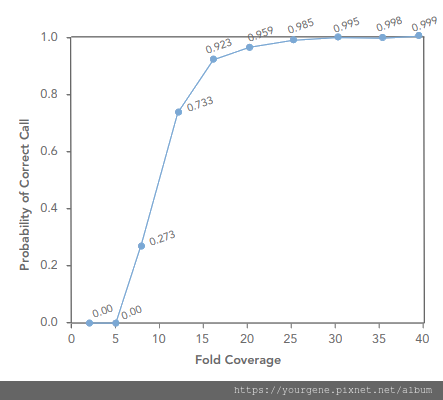
X軸:定序深度;Y軸:正確偵測異型合子出現單核苷酸多型性的機率。單核苷酸多型性:Single Nucleotide Polymorphism; SNP。(圖片來源:Ilumina®, Inc. Illumina Technical Note: Systems and Software.)
可是問題來了,假如我們是針對人類的全基因體進行定序,定序深度30倍,需要定序的量就是3G(人類染色體總長度)*30(定序深度)=90G(定序量),定序價格一般會落在台幣5萬元左右。如果您手握龐大經費,這價格對您來說,可能不是問題。但若您像作者一樣家徒四壁、月底吃土,那這筆定序費用就會是個沈重的負擔。更甚者,萬一您志向遠大,希望將研究範圍從單一個體拓展到群體層面,那您所需要的經費將會是5萬*定序樣品數=$$$$…元。
因此當我們需要利用有限經費去進行大規模定序,以利發表更高點數的paper,好早點拿到畢業證書或教授升等,目前有三種常見方法可以達到這個目的,那就是RAD-Seq、Pool-Seq、以及lcWGS(Low-coverage WGS;也有人稱之為"Low-pass" WGS),詳見圖二2。
第一種方法的RAD-Seq (Restriction-site Associated DNA Sequence)3,4,5,是利用限制性內切酶將樣本的基因體DNA先進行剪切,然後對被切下來的DNA片段進行「特定長度DNA片段(Restriction Associated DNA tag; RAD-tag)」的定序,確認被切下來的DNA片段都帶有這個可作為生物標記用的RAD-tag序列,以利後續的族群遺傳分析。理想上,由於限制性內切酶的專一性,我們可以讓來自相同物種的多個樣本,在每做一次酵素處理後,都能各被切出一樣的RAD-tag組合。如下圖二A所示,當我們利用RAD-Seq技術針對來自相同物種的五個樣品組合(棕、紫、綠、黃、紅)進行定序時,我們藉由酵素的專一性,讓這五個樣品都各被切出四個相同的序列區,然後進行定序。然而這個技術有兩個缺點:(1)酵素的處理有時候不會這麼完美;理論上這五個樣品的基因體DNA都能各被切出四個相同的RAD-tag片段作為生物標記之用,但實際上有些樣品能得到的RAD-tag卻可能少於四個,以致後續的分析出現問題。(2)不同樣品間基因表現的差異可能和RAD-tag的序列資訊無關,也就是說,真正會影響基因表現的染色體位點,其實是位於RAD-tag之外的序列區域上(如圖二A問號處所示)6,7。
圖二、RAD-seq (A)、Pool-seq (B)、lcWGS (C)示意圖
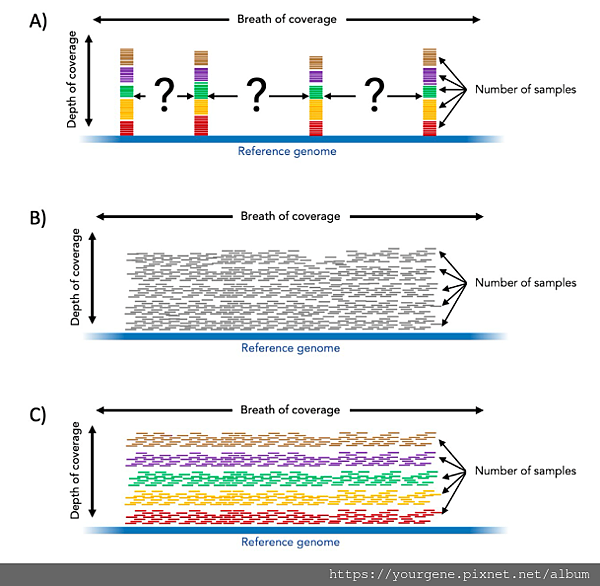
X軸:參考基因體的序列;Y軸:定序深度;上方標示:覆蓋範圍;右側標示:混合的樣品數。圖B中的序列因為無法區別五個樣品的定序結果,所以統一用灰色表示。(圖片來源:Lou, R.N., Jacobs, A., Wilder, A., & Therkildsen, N.O. Authorea. 2020 December 02.)
既然RAD-Seq具有上述缺陷,那全基因體定序就是改善上述問題的方法之一;然而標準全基因體定序的價格卻讓眾多研究者為之卻步。考慮價格問題的話,倒是有個替代方案,就是將想要定序的樣品先混合一起然後再去定序,就是所謂的Pool-Seq8,9。只要我們用來混合的「樣品數量」夠多,且Pool-Seq的「定序深度」夠深(如本文一開頭提到的30倍),就有機會可以取得「族群」特有的遺傳資訊,如上圖二B所示。換句話說,只要在Pool-Seq的分析結果中觀察到基因體序列某個位置上有「一致性」的染色體變異,就代表這些樣品都帶有這個染色體變異,進而獲得這個「族群」的遺傳資訊。然而,這個方法成也pool敗也pool;當我們將所有樣品都混合一起的同時,等於也把每個樣品的個別資訊給抹去了,如此一來就無法再從Pool-Seq的資料去進一步針對個別樣品進行研究或分類了。
正當RAD-Seq 和 Pool-Seq對於誰才是王道而吵得不可開交之際,lcWGS跳出來說話了:「爭什麼,不會摻在一起做成撒尿牛丸嗎?笨蛋!」。而lcWGS算是在RAD-Seq和Pool-Seq之間取得平衡點的一個方法,它將Pool-Seq的流程稍做了調整,改成先將每個樣品各自建庫(library preparation)後再去進行定序。只是這個作法所需要的定序深度(定序讀數reads)極低(大約在0.5倍~4倍左右),因此在降低定序成本的同時,仍能保有各自樣品的資訊。但是由於每個樣品的定序量實在是太低了,可想而知要獲得各個樣品「精確的」遺傳資訊其實是有困難的10,11。不過,這樣的結果對於解決「族群」的遺傳表現問題(例如:allele frequencies, linkage disequilibrium (LD) patterns),已經夠用了。至於lcWGS所提供的分析結果能有多準確呢?我們可以從圖三的「單核苷酸多型性(SNP)出現頻率」的估算結果窺知一二。
圖三、樣本數量與定序深度對「準確偵測SNP出現頻率」的影響
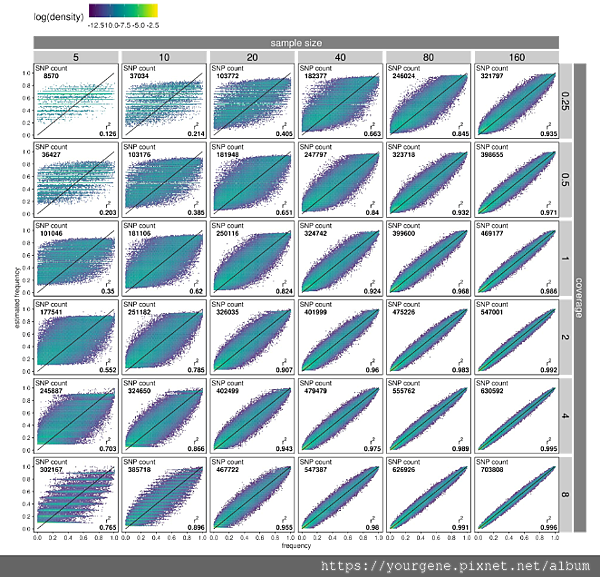
SNP:Single Nucleotide Polymorphism單核苷酸多型性。X軸代表SNP的實際出現頻率,左側Y軸代表lcWGS 對SNP出現頻率的估計結果。右側Y軸為定序深度。(圖片來源:Lou, R.N., Jacobs, A., Wilder, A., & Therkildsen, N.O. Authorea. 2020 December 02.)
如圖三所示,當樣本數量夠多(達160個),且定序深度夠深(每個樣品有8X)時,兩者對準確偵測SNP出現頻率的總影響(圖中的r2數值)可以高達0.996;換句話說,能準確偵測的機率可達99.6%。縱使定序深度下調至0.5X,只要樣本數量還有80個,r2數值依然可以維持在0.932。
「族群」的遺傳分析一直是很熱門的研究方向,不管是在基礎研究或是遺傳疾病的研究上。若能善用low-coverage WGS (low-pass WGS)相信應該可以讓您的研究結案報告更容易high-pass!
參考文獻
1. Ilumina®, Inc. Calling Sequencing SNPs. Illumina Technical Note: Systems and Software. Retrieved from https://www.illumina.com/Documents/products/technotes/technote_snp_caller_sequencing.pdf
2. Lou, R.N., Jacobs, A., Wilder, A., & Therkildsen, N.O. A beginner's guide to low-coverage whole genome sequencing for population genomics. Authorea. 2020 December 02. Retrieved from https://doi.org/10.22541/au.160689616.68843086/v2
3. Andrews, K.R., Good, J.M., Miller, M.R., Luikart, G., & Hohenlohe, P.A. Harnessing the power of RADseq for ecological and evolutionary genomics. Nature Reviews Genetics. 2016 Feb;17(2):81-92. Retrieved from https://doi.org/10.1038/nrg.2015.28
4. Davey, J.W., Hohenlohe, P.A., Etter, P.D., Boone, J.Q., Catchen, J.M., & Blaxter, M.L. Genome-wide genetic marker discovery and genotyping using next-generation sequencing. Nature Reviews Genetics. 2011 Jun;12(7):499-510. Retrieved from https://doi.org/10.1038/nrg3012
5. McKinney, G. J., Larson, W. A., Seeb, L. W., & Seeb, J. E. RAD seq provides unprecedented insights into molecular ecology and evolutionary genetics: comment on Breaking RAD by Lowry et al. Molecular Ecology Resources. 2016 Dec;17(3):356-361. Retrieved from https://doi.org/10.1111/1755-0998.12649
6. Tiffin, P., & Ross-Ibarra, J. Advances and limits of using population genetics to understand local adaptation. Trends in Ecology & Evolution. 2014 Dec;29(12):673-680. Retrieved from https://doi.org/10.1016/j.tree.2014.10.004
7. Lowry, D.B., Hoban, S., Kelley, J.L., Lotterhos, K.E., Reed, L.K., Antolin, M.F., & Storfer, A. Breaking RAD: An evaluation of the utility of restriction site‐associated DNA sequencing for genome scans of adaptation. Molecular Ecology Resources. 2017 Mar;17(2):142-152. Retrieved from https://doi.org/10.1111/1755-0998.12635
8. Schlötterer, C., Tobler, R., Kofler, R., & Nolte, V. Sequencing pools of individuals—mining genome-wide polymorphism data without big funding. Nature Reviews Genetics. 2014 Nov;15(11):749-763. Retrieved from https://doi.org/10.1038/nrg3803
9. Anderson, E.C., Skaug, H.J., & Barshis, D.J. Next‐generation sequencing for molecular ecology: a caveat regarding pooled samples. Molecular Ecology. 2013 Dec;23(3):502-512. Retrieved from https://doi.org/10.1111/mec.12609
10. Nielsen, R., Paul, J.S., Albrechtsen, A., & Song, Y.S. Genotype and SNP calling from next-generation sequencing data. Nature Reviews Genetics. 2011 Jun;12(6):443-451. Retrieved from https://doi.org/10.1038/nrg2986
11. Nielsen, R., Korneliussen, T., Albrechtsen, A., Li, Y., & Wang, J. SNP calling, genotype calling, and sample allele frequency estimation from New-Generation Sequencing data. 2012 Jul;PloS One. 7(7):e37558-e37568. Retrieved from https://doi.org/10.1371/journal.pone.0037558
12. GIPHY. You Shall Not Pass. Lord of the Rings gif. Retrieved from http://gph.is/1bgR8Yz





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
