作者:張家豪/有勁基因
有勁部落格文章《Patterned Flowcell平台的重複讀取問題》中曾提到:HiSeq 4000平台需要尋找文庫(library)的最佳上機濃度進行定序,不然會容易出現資料「重複讀取(duplicate reads)」的情況。那麼有沒有工具可以計算出定序資料中duplicate reads的實際比例並且將之移除呢?答案是:有的。本文會使用表一中的定序資料來為大家介紹兩種可供利用的軟體工具⼀FastQC以及fastp。
首先來介紹下表一定序示範資料的背景概況。duplicate_R1.fastq和duplicate_R2.fastq兩個定序檔案是「雙端(paired-end)定序資料」備註1,共有30條序列 (表一中的read1到reads15)。筆者特意為30條定序資料設計了下表中(A)到(E)五種不同的重複讀取狀況,以便介紹FastQC以及fastp兩種軟體工具的計算方式及進行效能的評估(詳見表一與圖一)。
表一、本文定序示範資料簡介
|
測試樣品名稱 |
R1:duplicate_R1.fastq |
R2:duplicate_R2.fastq |
|
定序資料總數 |
30條定序資料 |
30條定序資料 |
|
(A)R1&R2樣品中相同序列各讀取了2次:見序列read 14和read 15 |
序列整組相同 |
序列整組相同 |
|
(B)R1&R2樣品中相同序列各讀取了3次:見序列read 9, read 10, read 13 |
序列整組相同 |
序列整組相同 |
|
(C)R1&R2樣品中相同序列各讀取了4次:見序列read 7, read 8, read 11, read 12 |
序列整組相同 |
序列整組相同 |
|
(D)R1&R2樣品中相同序列各讀取了2次:見序列read 2和read 3 |
序列只有前12bp相同 |
序列只有前32bp相同 |
|
(E)R1&R2樣品中相同序列各讀取了2次:R1: 序列read 5和read 6 / R2: 序列 read 1和read 4 |
序列只有前50bp相同 |
序列只有前50bp相同 |
圖一、定序資料中特意設計了5種不同的重複讀取狀況
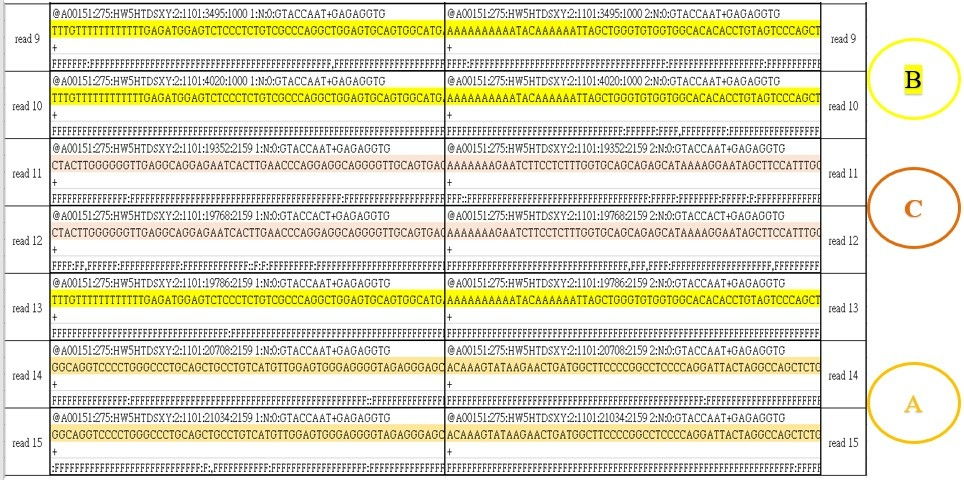
(A)R1&R2樣品中相同序列各讀取了2次,序列整組相同;(B)R1&R2樣品中相同序列各讀取了3次,序列整組相同;(C) R1&R2樣品中相同序列各讀取了4次,序列整組相同;(D) R1&R2樣品中相同序列各讀取了2次,序列只有前12bp相同;(E) R1&R2樣品中相同序列各讀取了2次,序列只有前50bp相同。(圖片來源:張家豪/有勁基因)
工具一、FastQC軟體(版本:v0.10.1, 2012年3月)
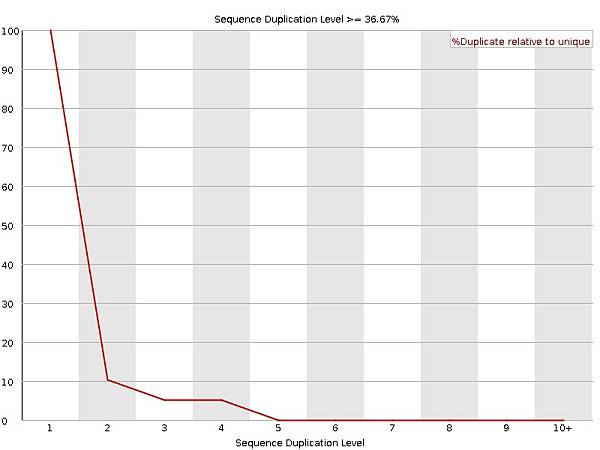
FastQC1是評估定序品質最常見的軟體,由於這個軟體一次只能計算其中一個定序資料的duplicate reads比例(例如:duplicate_R1.fastq 或 duplicate_R2.fastq其一),且只取序列中前50個base pair進行計算,因此duplicate reads的比例容易偏高。例如duplicate_R1.fastq定序檔案的duplicate reads比例計算結果就高達36.67%(見圖二)。
FastQC duplication=所有重複讀取的序列/定序資料總數=[11=(A+B+C+E)]/30
(D的重複讀取狀況中,R1序列只有前12bp相同,R2只有前32bp相同,所以不計入;至少要前50bp相同才會計入。)
圖二、FastQC軟體工具的duplicated reads比例計算結果

圖片來源:張家豪/有勁基因
工具二、fastp軟體(最新版本:v0.22.0, 2021年8月)2
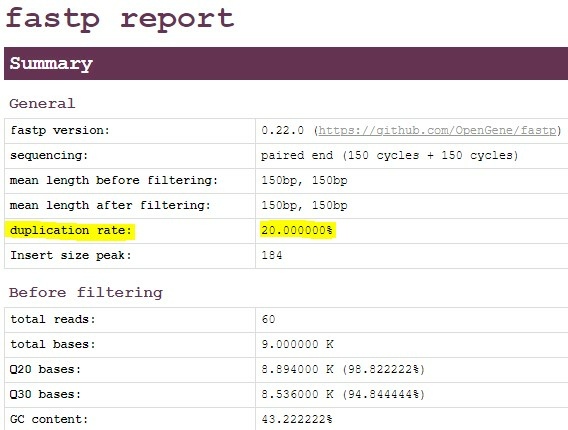
fastp軟體於2018年推出3,能夠快速分析與處理雙端定序資料,軟體功能強大。今年8月熱騰騰剛出爐的最新版本,除了改善了先前版本中duplicate reads的計算方式 備註2 ,還額外增加了移除duplicate reads的功能。示範檔案的duplicate reads比例計算結果為20%(見圖三)。
fastp duplication=1-[特殊讀取的序列Unique Reads/定序資料總數]
=1-{[30-1(A)-2(B)-3(C)]/30}=6/30=20%

圖三、使用fastp軟體工具的duplicate reads比例計算結果

圖片來源:張家豪/有勁基因
由上可見,每個軟體所提供duplicate reads比例計算結果都有所不同;此外,隨著軟體版本的更新,也需要去瞭解更新前後的版本是否有所差異。最後,筆者擔心讀者還是不清楚這兩個軟體之間的差異,所以整理了下面的比較表格提供大家參考。
表二、不同軟體的duplicate reads計算方式比較
|
軟體 |
duplicate reads的計算方式 |
duplicate reads的移除 |
|
FastQC (版本:0.10.1) |
1、只能計算一個定序資料 |
無此功能 |
|
fastp (舊版:0.20.1) |
1、能計算雙端定序資料 |
無此功能 |
|
fastp (新版:0.22.0) |
1、能計算雙端定序資料 |
有此功能 |
表格來源:張家豪/有勁基因
【備註1】測試的定序資料已上載在Github上,供讀者們下載。
網址: https://github.com/jiahao0610/duplication
【備註2】筆者實際測試過舊版fastp軟體後,除了R1序列取前12bp,R2序列取前32bp作duplicate reads計算之外,還會有其他因子影響duplication reads比例的計算,如N base的有無,因此建議使用新版fastp軟體分析較佳。
參考文獻
1、Simon Andrews. (2021). FastQC. Retrieved from https://github.com/s-andrews/FastQC
2、OpenGene. (2020). fastp. Retrieved from https://github.com/OpenGene/fastp
3、Shifu Chen, Yanqing Zhou, Yaru Chen, Jia Gu. fastp: an ultra-fast all-in-one FASTQ preprocessor. Bioinformatics. 2018 Sep; Volume 34(17):i884-i890. Retrieved from https://doi.org/10.1093/bioinformatics/bty560





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
