 作者:黃千容/有勁基因
作者:黃千容/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(1,461)
 作者:張家豪/有勁基因
作者:張家豪/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(1,281)
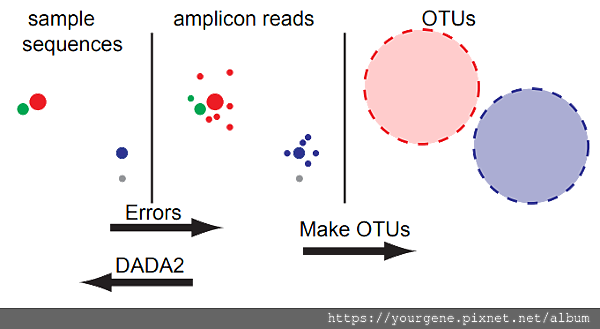 作者:張益祥/有勁基因
作者:張益祥/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(2,744)
 作者:張家豪/有勁基因
作者:張家豪/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(1,198)
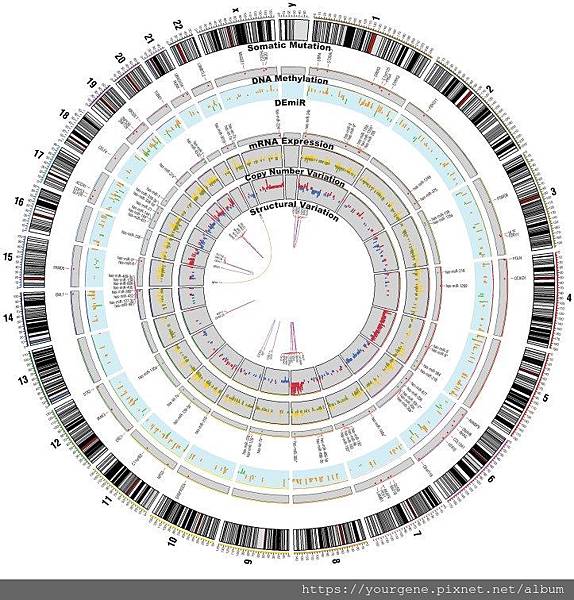 作者:張家豪/有勁基因
作者:張家豪/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(1,969)
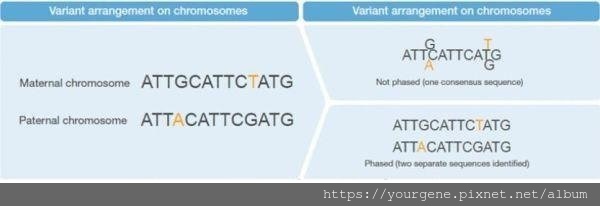 作者:張益祥/有勁基因
作者:張益祥/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(810)
 作者:鄭翰欽/有勁基因
作者:鄭翰欽/有勁基因 TIGS 發表在 痞客邦 留言(0) 人氣(860)
 作者:張益祥/有勁生物科技
作者:張益祥/有勁生物科技 TIGS 發表在 痞客邦 留言(0) 人氣(1,775)
 作者:鄭翰欽/有勁生物科技
作者:鄭翰欽/有勁生物科技 TIGS 發表在 痞客邦 留言(0) 人氣(1,005)
 作者:王育彬/有勁生物科技
作者:王育彬/有勁生物科技 TIGS 發表在 痞客邦 留言(0) 人氣(728)










