作者:葉佳妤/有勁基因
DNA甲基化(DNA methylation)是指DNA分子上的甲基化修飾。原核生物的甲基化修飾有助於DNA複製之後對錯誤配對的修補(Mismatch Repair; MMR);其經常發生在5′-GATC-3′序列中腺嘌呤(adenosine; A)的N6分子上,藉由產生AN6-CH3來區分新舊股DNA,舊股DNA上有AN6-CH3修飾而新股無,於是內切酶便能正確切除新股上的合成錯誤,進行DNA的修補。此外,若5′-GATC-3′序列上具有AN6-CH3的甲基化修飾也能輔助原核生物的限制修飾系統(restriction modification system)去抵禦外來噬菌體的入侵。真核生物的甲基化修飾則常發生在胞嘧啶(cytosine; C)的5號C上,將其轉化為5′-甲基胞嘧啶(5′-methylcytosine; 5mC)。而在哺乳動物身上,DNA序列的某些特定區域,例如啟動子區域(promoter region)、第一外顯子(first exon)等都是常見的CpG雙核苷酸聚集區域(因此又稱之為CpG島; CpG island),這些區域上如果發生了甲基化修飾,一般來說,都和基因調控脫不了關係。
DNA的甲基化修飾能在不改變DNA序列的情況下改變基因的表現,屬於表觀遺傳學(epigenetics)的一種基因調控機制。近年來,愈來愈多研究指出曾在不同類型癌症病人血液中的游離DNA(cell-free DNA; cfDNA)上發現異常甲基化的情形。cfDNA的甲基化目前被認為可用來作為癌症診斷、預後及預測治療後反應的生物標記,因此這方面的研究愈來愈受到矚目。
那麼,cfDNA的甲基化要如何定量呢?由於cfDNA在血漿或體液中含量稀少,而且容易降解並易被基因體DNA(genomic DNA; gDNA)所汙染,想要做到高敏感又具高特異性的cfDNA甲基化定量有些訣竅。本文將介紹針對DNA甲基化修飾序列所進行的定量聚合酶鏈鎖反應─「定量甲基化特異性PCR (qMSP; Quantitative Methylation-Specific Polymerase Chain Reaction)」1;這個技術簡單、快速又符合成本效益,可以用來研究癌症病人cfDNA的甲基化修飾。
qMSP的大致流程為:血液抽取後放入含有K2ETDA註1抗凝血劑的採血管或專門用來採集、保存cfDNA的採血管內,接著利用兩階段不同轉速的離心將血漿從血液中分離出來,然後萃取出血漿中的cfDNA。cfDNA濃度確認後,再對cfDNA進行變性(denature),將雙股打開成兩個單股,之後利用亞硫酸氫鈉(sodium bisulfite; NaHSO3)將DNA進行處理。DNA在經過處理後,其上的胞嘧啶(C)在正常情況下會轉變為尿嘧啶(U);但若胞嘧啶(C)的5號C被甲基化過(5mC),其鹼基形態則不會受亞硫酸氫鈉處理所影響,仍維持5mC。接著就是將引子(primer)黏合上去,進行甲基化特異性PCR(MSP; Methylation-Specific Polymerase Chain Reaction)(詳見圖一);在此同時,搭配SYBR Green染劑、或者專門黏合甲基化序列亦或是專門黏合未甲基化序列的TaqMan標記探針,即可針對甲基化或未甲基化的特定序列進行即時定量。換句話說,qMSP可說是MSP與Q-PCR(也就是「定量即時PCR(Quantitative Real-Time Polymerase Chain Reaction)」的技術結合。關於Q-PCR可參考有勁部落格文章《Q-PCR (Real-Time PCR)》。
圖一、有甲基化與未甲基化的序列,經過亞硫酸氫鈉及甲基化特異性PCR處理後的鹼基變化
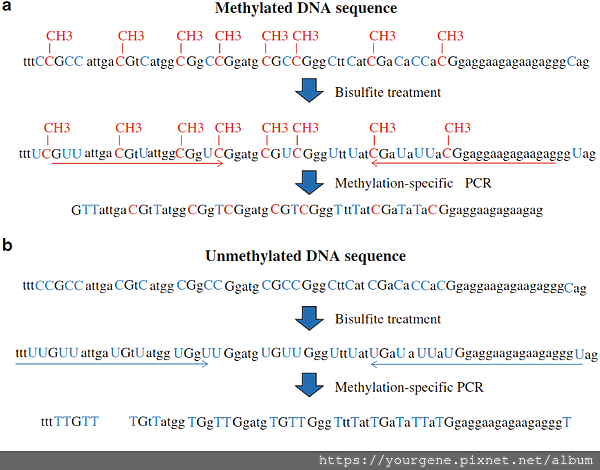
紅色標記為有甲基化的胞嘧啶鹼基,藍色標記為未甲基化的胞嘧啶鹼基。紅色及藍色左右橫向箭頭分別代表設計過的會針對甲基化及非甲基化鹼基做特異性黏合的引子對。DNA經過亞硫酸氫鈉處理後,未被甲基化過的C會轉變成U,之後在MSP反應過程中,U會再轉變為胸腺嘧啶(Thymine; T)。甲基化過的5mC則不會因為亞硫酸氫鈉處理而產生任何變化,依舊維持5mC,即便在MSP處理後,也仍維持原來C的鹼基形態。(圖片來源: Sigalotti, L. et al. (2019). Cell-free DNA as Diagnostic Markers (pp 137-162). (Methods in Molecular Biology, vol 1909). New York: Humana Press.)
qMSP能否成功,關鍵在於其引子的設計。人類基因約有6到7成的5′端啟動子區是CpG雙核苷酸聚集區域。此區域內的DNA序列在正常細胞中,常是未經甲基化的序列;若在此區域內發現甲基化過的序列,通常都和異常細胞的形成有關。為了研究癌症病人cfDNA的甲基化修飾,qMSP的引子通常都會拿此區域的DNA序列來做設計。如果想將qMSP引子設計地更準確、並根據qMSP結果評估DNA的甲基化程度,建議可以搭配使用MethPrimer註2 與NCBI-Gene 這些方便的生物資訊網站。
註1:K2EDTA是一種螯合劑,主要的作用就是能與鈣離子結合;鈣離子與K2EDTA結合後,凝血酶就無法作用,凝血機制便可被抑制。因此,K2EDTA在臨床檢驗上,常作抗凝血劑使用。
註2:MethPrimer是一個輔助DNA甲基化研究的網站,該平台提供許多研究DNA甲基化的免費工具和資料庫,包括預測基因可能的CpG島序列位置和設計qMSP引子的程式工具等等。
參考資料
1. Sigalotti, L. et al. (2019). Quantitative Methylation-Specific PCR: A Simple Method for Studying Epigenetic Modifications of Cell-Free DNA. In Cell-free DNA as Diagnostic Markers (pp 137-162). (Methods in Molecular Biology, vol 1909). New York: Humana Press. Retrieved from https://doi.org/10.1007/978-1-4939-8973-7_11





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
