作者:徐于晴/有勁生物科技
ENCODE是美國NHGRI (National Human Genome Research Institute)在2003所開始的一個計畫,目的是希望能註解人類基因體上的功能,包含去註解基因、RNA、轉錄調節相關的區域 (transcriptional regulatory regions)、染色質狀態(chromatin state)、DNA甲基化 (DNA methylation)等項目。下圖是ENCODE所想要註解的目標及其相對應的研究方法統整:
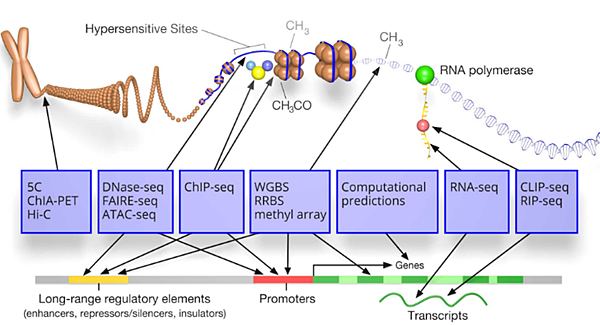
ENCODE在推行上分為兩大階段:
- Pilot project phase (2003-2007): 此階段以建立與測試實驗和分析流程為主,主要針對200筆資料中,分散於各處且總長約30Mb的人類基因體區域 (大約人類基因體的1%)來做分析。
- Production phase (2007-現在): 此階段使用前一階段所建立的實驗和分析流程,將分析目標拓展到整個人類基因體。
除了人類基因體的註解之外,ENCODE所建立的實驗及分析流程也被其他計畫用來註解其他種生物的基因體,像是老鼠、果蠅、線蟲等模式生物,其相關的計畫有:
Mouse ENCODE:註解老鼠的基因體
modENCODE:註解果蠅、線蟲的基因體
這些計畫的資料也都可以在ENCODE官網上的data portal找得到:https://www.encodeproject.org/matrix/?type=Experiment
ENCODE資料庫中包含有原始實驗產生的檔案 (raw data)及有分析處理過的檔案 (processed data),常見的檔案格式有FASTQ、BAM、bigWig、bigBed等,後兩種檔案格式主要是為了和UCSC genome browser整合,將ENCODE資料圖形化呈現的檔案格式。目前ENCODE資料在它的官網、NCBI GEO、Ensembl、UCSC等地方都可以下載,以從ENCODE官網下載data為例:
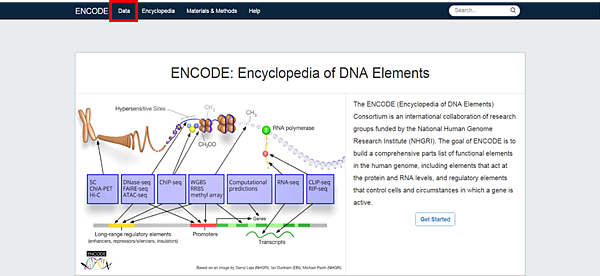
1. 在ENCODE的官網首頁,左上角會有Data的選項:
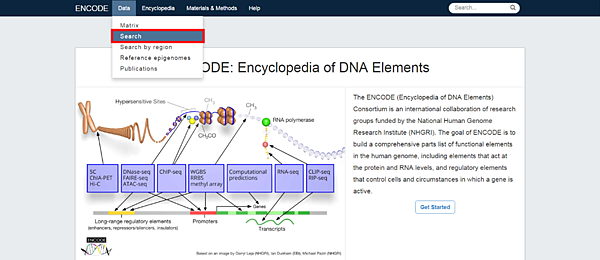
2. 點下去之後再選擇 Search:
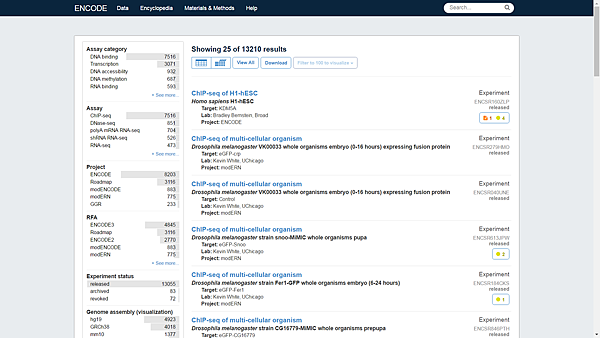
3. 就可以進到data搜尋的頁面,此頁面的左側有可以用來篩選想要的data的篩選條件,包含有實驗方法、物種、定序類型、細胞種類等:
4. 點選任一個dataset,可進到該dataset的資訊頁面,例如:
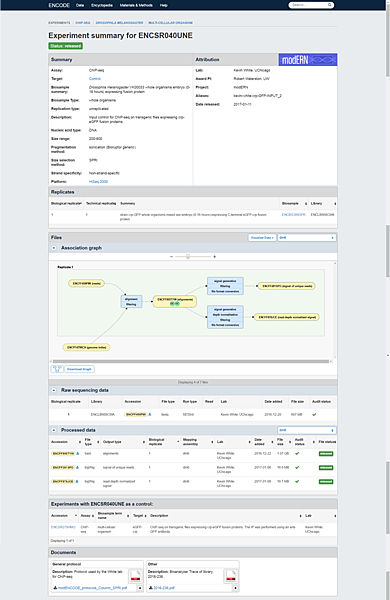
Summary的部分會有dataset的相關資訊,raw sequencing data為原始定序出來的檔案,processed data為分析過的檔案,只要按acession旁邊的下載符號就可以下載,最下面的documents則為實驗和分析流程的詳細說明文件。
除了直接點官網上的下載連結外,也可以利用程式批次抓取檔案,大致的流程如下:
1. 先利用程式透過REST API抓取網頁上的資訊,下載下來的檔案會是JSON的格式,如下圖:

2. 在以上的資料中,抓取出”href”的欄位,例如下圖中反藍的部分:

反藍為"href": "/files/ENCFF769WTD/@@download/ENCFF769WTD.fastq.gz",這是原本網頁中的其中一個data的位置,只要在前面加上https://www.encodeproject.org,就是這個data的下載連結。
3. 利用以上所得到的下載連結,就可以在server下載檔案:
可以用wget的指令,例如:
wget https://www.encodeproject.org/files/ENCFF769WTD/@@download/ENCFF769WTD.fastq.gz
或是用curl的指令,例如:
curl -O -L https://www.encodeproject.org/files/ENCFF769WTD/@@download/ENCFF769WTD.fastq.gz
更多關於資料下載的詳細說明和Python的範例程式碼可以在以下網頁找到:
https://www.encodeproject.org/help/rest-api/
參考資料:
https://www.encodeproject.org/




 留言列表
留言列表 {{ article.title }}
{{ article.title }}
