作者:鄭翰欽/有勁基因
人類基因約有2萬個;這些基因不但會彼此調控還會互相影響。某個基因一旦發生突變,依據突變的嚴重程度與突變基因的種類,對個體會造成不同程度的影響。像緘默突變(silent mutation; synonymous mutation)這類的輕微變異,因為對其所轉譯的胺基酸序列不造成影響,所以並不會改變所製造出來的蛋白質功能。而像移碼突變(frameshift mutation)這類非常嚴重的突變,突變位點之後的胺基酸序列會全部受到影響,以致製造出來的蛋白質因結構改變而失去功能,就會影響個體的生理狀況甚至引發疾病。
於是有些研究機構就去收集歸納已知的「基因變異」與疾病之間的關係,將之整理成比對參考用的資料庫,例如:ClinVar1與COSMIC2等等資料庫。然而基因變異與疾病關聯性的這些資料是透過研究與收集得來的,多少都會有一些罕見變異因未被收錄在資料庫而被遺漏掉,這樣的話,該如何才能知道這些罕見變異與疾病之間的關聯呢?此時就必須從「基因本身」來找線索,查詢這個基因的功能以及和該基因有關的疾病,然後再比對該基因的罕見變異對疾病相關的蛋白質序列所造成的影響,如此才能找出關聯性。
哪裡可以找到有收集「基因功能」與疾病關聯的資料庫呢?講到這個就一定要提到人類表型功能分類資料庫(The Human Phenotype Ontology;簡稱HPO)3。HPO採用類似基因功能分類資料庫(Gene Ontology;簡稱GO)的資料結構;每一個HPO索引裡的項目「term」就代表人類疾病的生理表型。如圖一所示的HPO term說明頁,描述了生理表型的功能定義與分類、相關疾病編號與疾病名稱、以及與此表型功能有關的基因等等。
圖一、HPO term說明頁面的介紹
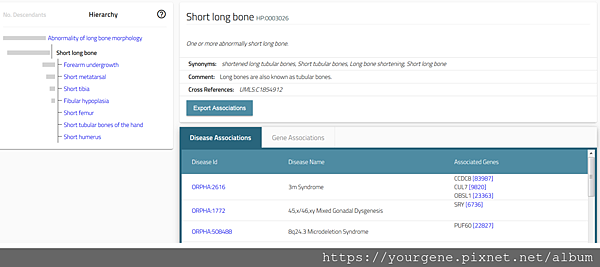
左側為HPO term的分類層級;所選擇的HPO term會以黑色粗體標示,同時也會列出其父層與子層的HPO名稱。右側則為所選擇的HPO term說明,會表列出生理表型的功能定義與分類、相關疾病編號與疾病名稱、以及與此表型功能有關的基因等資訊。(圖片來源:Köhler, S., et al. Nucleic Acids Research. 2019 Jan; 47(D1):D1018-D1027.)
那麼,這個HPO「基因功能」與疾病關聯的資料要如何才能與「基因變異」串連起來呢?舉例來說,某患者出現類似玻璃娃娃(成骨不全症;Osteogenesis Imperfecta)的疾病表徵,我們懷疑這可能是患者體內某些基因位點發生突變所造成,但是要如何才能知道是哪些基因發生了變異呢?此時,我們會依循以下流程去尋找可能的基因變異位點。
1. 首先,先在HPO網站輸入疾病的名稱;資料庫可能會提供很多結果,請選擇最符合患者狀況的項目(如圖二所示)。
2. 在疾病的頁面中,會列出對應的HPO term(圖三HPO Associations);此時可以先選擇符合的HPO term,再去挑選符合的候選基因(圖四)。有些情況下,所選的疾病在資料庫中就有充分的證據指出其與某特定基因有關,此時網頁就會列出對應的基因(圖三Gene Associations),我們就可以直接針對這些基因去做後續的判讀。
3. 然後,再回到實驗室從檢測結果去確認資料庫所給的基因有沒有發生變異。基因若有突變,就仔細去比較發生變異的基因型(genotype)、突變的影響程度(例如:移碼突變或是緘默突變),並由此來評估該變異會不會是造成此疾病的原因。
圖二、在HPO網站以Osteogenesis Imperfecta做為關鍵字所搜尋到的結果
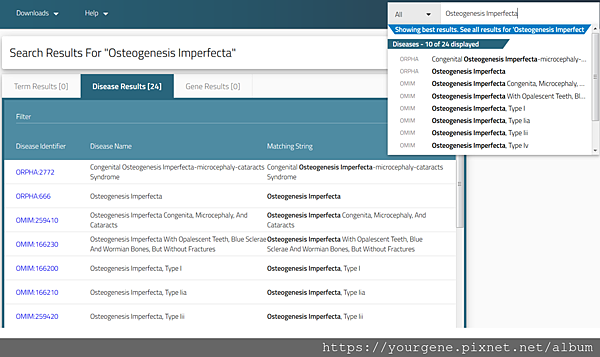
(圖片來源:Köhler, S., et al. Nucleic Acids Research. 2019 Jan; 47(D1):D1018-D1027.)
圖三、疾病的說明頁
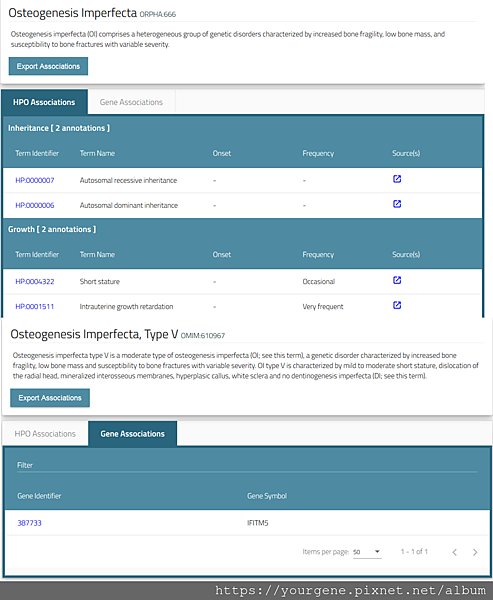
HPO資料庫在疾病的說明頁面中會說明這個疾病的表徵、其HPO term (HPO Associations)。假如這個疾病已經有充分證據指出其與哪個特定基因有關,那麼在Gene Associations的標籤中就會列出相關的基因。
(圖片來源:Köhler, S., et al. Nucleic Acids Research. 2019 Jan; 47(D1):D1018-D1027.)
圖四、與 HPO term有關聯的基因列表
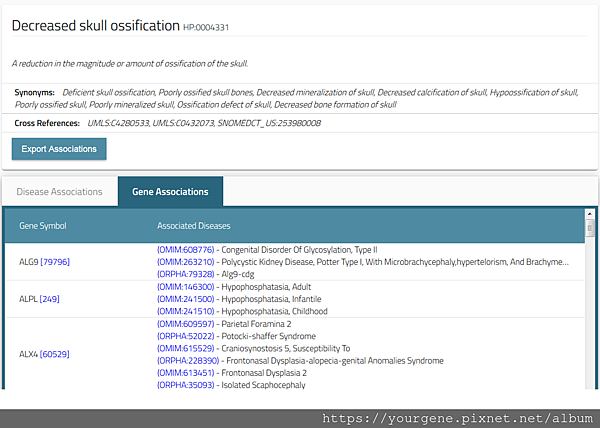
(圖片來源:Köhler, S., et al. Nucleic Acids Research. 2019 Jan; 47(D1):D1018-D1027.)
看到這邊,讀者或許會問:「這樣做就算找到了基因,也沒辦法確定疾病就是這個基因發生變異所造成的呀?」。確實,透過這個方法所找到的結果無法當作直接證據,只能作為致病可能原因的參考。但是,之所以會採用這個方法,是因為在ClinVar或COSMIC資料庫找不到對應的資訊,所以只好先採取這種迂迴方式,先將基因變異與疾病基因之間的關聯連建立起來,然後再將結果交給醫療研究團隊做更進一步的研究確認。一旦確認,未來這資訊就有可能被收錄到資料庫中,提供使用者更多的參考。
參考文獻
1. Landrum, M.J., et al. ClinVar: public archive of relationships among sequence variation and human phenotype. Nucleic Acids Research. 2014 Jan; 42(D1):D980-D985. https://doi.org/10.1093/nar/gkt1113
2. TATE, J.G., et al. COSMIC: the catalogue of somatic mutations in cancer. Nucleic Acids Research. 2019 Jan; 47(D1):D941-D947. https://doi.org/10.1093/nar/gky1015
3. Köhler, S., et al. Expansion of the Human Phenotype Ontology (HPO) knowledge base and resources. Nucleic Acids Research. 2019 Jan; 47(D1):D1018-D1027. https://doi.org/10.1093/nar/gky1105





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
