在Re-sequencing 的分析中,將Paired-end reads 對回到參考序列後的SAM 格式中,其中一個欄位”FLAG”,將會記錄paired-end reads 對回參考序列的特性。FLAG定義reads對回參考序列後的幾種特性,如圖一所式,其計算方法則是將read含有的每一個特性所對應的數值相加。

圖一:SAM format中的FLAG欄位
藉由以上的定義,如果一對paired-end reads 在合理的inserted-size下map到同一條參考序列,稱為Concordant pairs,會有圖二及圖三的兩種情形。
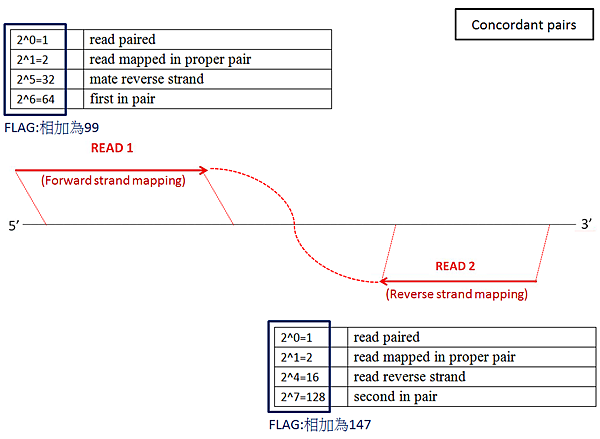
圖二: Concordant pairs 對回參考序列的情形,其READ1為forward mapping,FLAG為99;READ2為reverse mapping,FLAG為147。

圖三: Concordant pairs 對回參考序列的情形,其READ1為reverse mapping,FLAG為83;READ2為forward mapping,FLAG為163。
若一對paired-end reads的READ1及READ2分別map到不同的參考序列,稱為Discordant pairs,有圖四及圖五的兩種情形。在分析的應用上,如是genomic DNA,有可能是genome rearrangements後造成,或病毒序列鑲嵌到宿主DNA後造成的情形。如是RNA-Seq,則有可能fusion gene或trans-splicing造成的。

圖四: Discordant pairs 對回參考序列的情形,其READ1為forward mapping,FLAG為97;READ2為reverse mapping,FLAG為145。
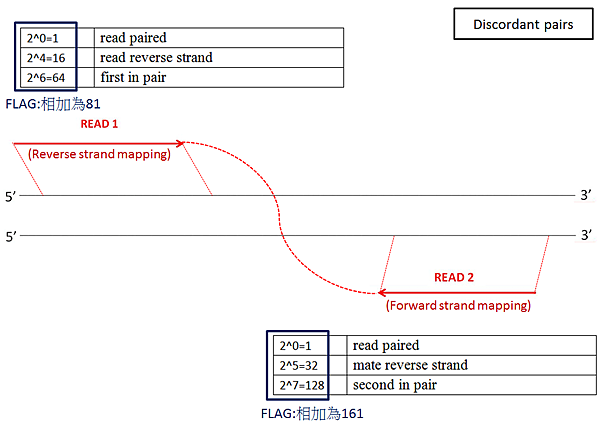
圖五: Discordant pairs 對回參考序列的情形,其READ1為reverse mapping,FLAG為81;READ2為forward mapping,FLAG為161。
若一對paired-end reads的READ1及READ2只有其中一邊有map到參考序列,另一邊的READ沒辦法對到參考序列,則有圖六~圖九的情形,unmapped read依造定序的種類有不同的可能,如是genomic DNA,unmapped read有可能是橫跨genome rearrangements中的break point,或是病毒序列鑲嵌到宿主DNA後的Insertion size。如是RNA-Seq,則有可能是novel gene中的exon boundary或fusion gene或trans-splicing中的fusion transcript的break point。
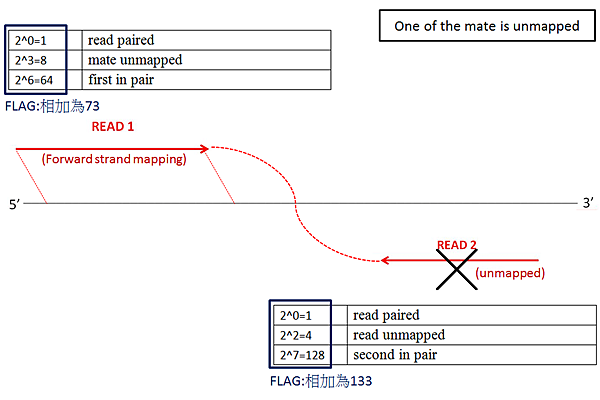
圖六: READ1為forward mapping,其FLAG為73;READ2為unmapped,FLAG為133。

圖七: READ1為reverse mapping,其FLAG為89;READ2為unmapped,FLAG為133。

圖八:READ1為unmapped,FLAG為69;READ2為forward mapping,FLAG為137。

圖九: READ1為unmapped,FLAG為69;READ2為reverse mapping,FLAG為153。
這邊我們舉了Illumina platform定序後的paired-end reads常碰到的flag組合,在不同sequencing platform或alignment parameters下會有很多可能性,可供後續生物資訊分析來擷取資訊。
補充:在 2013年5月22日,本部落格發表一篇對於FLAG換算的文章,請參考"SAM format中的FLAG應用 - FLAG換算"





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
