作者:黃勤方/有勁生物科技
癌症免疫治療目的是透過疫苗去啟動癌症的免疫週期,希望藉由活化患者的T細胞去正確分辨腫瘤細胞與正常細胞,讓免疫系統只針對腫瘤細胞進行毒殺,能更有效專一地控制腫瘤的生長與惡化。腫瘤的發生與基因突變有關;引發與維持腫瘤生長存活的「大哥級」關鍵性突變─driver mutation(驅動突變),以及其他看似不太重要的「跟班級」突變─passenger mutation(附隨突變),不論是哪一種,都有可能改變蛋白質序列。從免疫的角度來看,也就是說,基因突變有可能讓腫瘤細胞表現出正常細胞所沒有的蛋白─新生抗原(neoantigen),而該蛋白上決定抗原特異性的胜肽區域即為新生抗原決定位(neoepitope)。1如何利用找出患者癌細胞的基因突變位點、以及具特異性的新生抗原決定位,來正確預測並選出合適的新生抗原標的,然後設計出個人化的癌症疫苗,是當今醫學界的新挑戰。
癌症免疫治療在個人化疫苗的設計方面,可以利用NGS技術找出癌症患者的基因體突變位點。其臨床標準作業程序上,通常會利用常規檢查或手術時採集腫瘤組織,冷凍保存新鮮組織檢體,或是用福馬林固定石蠟來包埋組織檢體。同時也會抽血,將血球細胞當作患者的健康組織對照組,以避免將生殖細胞的遺傳變異(germline variants)位點誤認為癌細胞突變的位點。此外還會萃取腫瘤組織和血球細胞兩種檢體的核酸,並進行DNA外顯子序列分析(DNA exome-sequencing);腫瘤細胞則還需額外執行mRNA序列分析。之後,再進行定序數據的演算分析,找出人類MHC基因─HLA序列(見文末「免疫學小筆記」的說明)中的單一核苷酸變異(single nucleotide variations, SNV)位置。由於腫瘤細胞的基因突變多是以單一位點突變為主,因此一旦在基因表現區域內發現SNV,其所表現出的異常蛋白就會被列入新抗原決定位的候選名單。候選名單出爐後,需先評估各候選蛋白與MHC結合的親和力高下、以及結合之後引發T細胞活化的能力如何,然後才進行疫苗的設計與製備。最後才能將試驗疫苗注入患者體內進行免疫治療的臨床測試。2(詳見圖一)
圖一、尋找癌症新生抗原與個人化癌症疫苗設計的流程圖
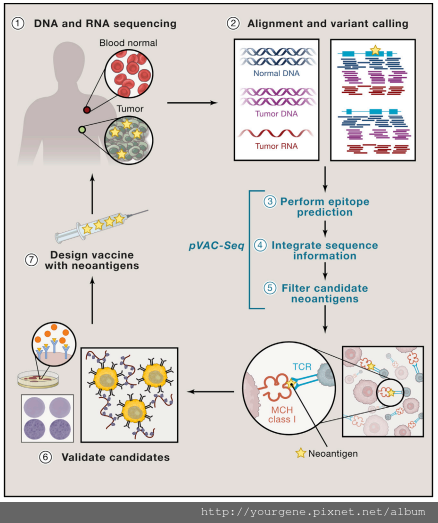
圖片來源:Liu, X.S. and Mardis, E.R. Cell. 2017 Feb 9; 168(4), 600-612. (註:圖片中的MCH class I為期刊誤植,正確應為MHC class I)
由於NGS定序結果的資料量相當龐大,後續資料分析既要找出變異處又要預測醣蛋白之間的結合能力,過程繁瑣,因此需要許多分析工具的輔助,並且得進行複雜參數的調整。Zlatko Trajanoski教授與其團隊將NGS在癌症免疫與免疫治療的資料探勘中可能會使用到的相關分析工具整合成一個名為TIminer (Tumor Immunology miner)的套件,讓大家在用pipeline分析NGS數據時能更容易更順利。3 套件中所使用的分析工具及相關架構如下圖二所示。
圖二、TIminer套件的分析流程架構
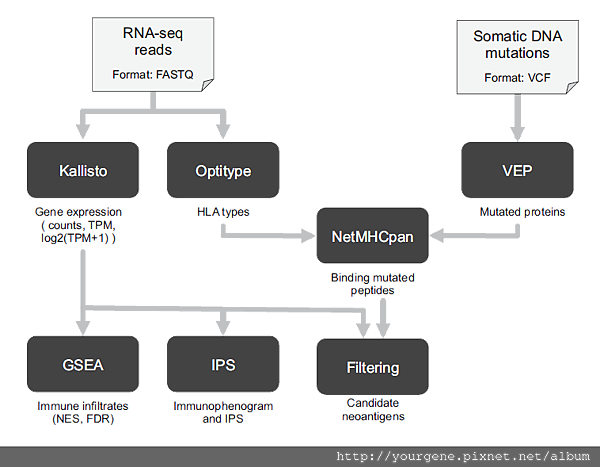

(圖片來源:Tappeiner, E., et al. 2017 Oct 1; 33(19), 3140–3141.)
本文簡易介紹了NGS在癌症疫苗設計過程中所扮演的角色。然而基因從複製到表現蛋白質的過程當中,有許多環節都可能對結果造成影響,而且這些影響能進一步引起的免疫反應也很複雜;因此,疫苗的開發其實比想像中更為困難。如果癌症是「24個比利」(出自1981年丹尼爾·凱斯描述多重人格患者的真人真事傳記小說<The Minds of Billy Milligan>),那麼NGS就好比是基因的心理測驗評量工具。透過NGS去了解基因、以及基因體突變與疾病之間的關係,有朝一日或許可以讓患者不再需要盲目地以身試藥而飽受副作用之苦,而能以更精確的判斷去為個別患者找出最合適的治療方式。
*免疫學小筆記:
MHC(major histocompatibility complex;主要組織相容性複體)是細胞表面上的醣蛋白分子,可與抗原結合,並將結合後的抗原形式呈現給T細胞。人類的MHC位於第6對染色體上,由於人類最先被發現的MHC蛋白是在白血球表面上,所以人類的MHC醣蛋白又被稱為人類白血球抗原群(human leukocyte antigens, HLA)。MHC具有多型性,在不同個體上的表現會有差異。MHC大致可分為MHC class I與MHC class II兩類。前者可將抗原呈現給 CD 8+ CDL T cell (細胞毒性T細胞;又稱殺手T細胞),其基因為HLA-A, HLA-B, HLA-C。後者可將抗原呈現給CD 4+ helper T cell (輔助T細胞),其基因為HLA-DQ, HLA-DR。
參考文獻
1.Sahin, U. & Türeci, Ö. Personalized vaccines for cancer immunotherapy. Science. 2018 Mar 23; 359(6382), 1355–1360.
https://doi.org/10.1126/science.aar7112
2.Liu, X.S. & Mardis, E.R. Applications of Immunogenomics to Cancer. Cell. 2017 Feb 9; 168(4), 600-612.
https://doi.org/10.1016/j.cell.2017.01.014
3.Tappeiner, E., Finotello, F., Charoentong, P., Mayer, C., Rieder, D., and Trajanoski, Z. (2017). TIminer: NGS data mining pipeline for cancer immunology and immunotherapy. Bioinformatics. 2017 Oct 1; 33(19), 3140–3141.
http://doi.org/10.1093/bioinformatics/btx377
4.Division of Bioinformatics Biocenter, Medical University of Innsbruck. (2016). TIminer: Analytical pipeline for mining tumor-immune cell interactions from genomic data. Retrieved from
http://www.icbi.at/software/timiner/timiner.shtml





 留言列表
留言列表 {{ article.title }}
{{ article.title }}
